

高等学校化学学报 ›› 2024, Vol. 45 ›› Issue (11): 20240311.doi: 10.7503/cjcu20240311
收稿日期:2024-06-26
出版日期:2024-11-10
发布日期:2024-08-07
通讯作者:
申华莉
E-mail:shenhuali@fudan.edu.cn
基金资助:Received:2024-06-26
Online:2024-11-10
Published:2024-08-07
Contact:
SHEN Huali
E-mail:shenhuali@fudan.edu.cn
Supported by:摘要:
在疾病发生过程中, 蛋白质的表达水平、 修饰状态或相互作用模式的变化有可能反映病理状态或疾病进展, 因此蛋白质常被用作疾病标志物. 基于质谱的蛋白质组学发现了大量的潜在疾病标志物, 这些标志物及其质谱检测方法能否进一步推广至临床检测, 定量准确性和稳定性是关键. 本文合成了32条标准肽段, 对数据非依赖性采集(DIA)、 多反应监测(MRM)及平行反应监测(PRM) 3种有潜力在临床质谱方面大规模应用的定量方法进行了比较和评估. 将肽段稀释成4个不同的浓度来模拟不同丰度的实际样本, 用以上3种质谱方法采集数据后, 比较了肽段发现率、 标准曲线线性、 日间和日内精密度及保留时间(RT)漂移等情况, 总结了3种定量方法各自的优缺点和潜在应用方向. 结果表明, MRM定量方法最适合临床应用, 在于其优异的日间日内稳定性、 灵敏度和定量精度; PRM适合科研方向靶向定量, 因其拥有最高灵敏度和较高稳定性定量精度; 而DIA由于高通量而依赖于软件和算法解析, 定量数目多的同时, 对于特定肽段定量准确性比PRM/MRM差.
中图分类号:
TrendMD:
张磊, 申华莉. 标志物研究中常用蛋白组学质谱方法的定量准确性评估. 高等学校化学学报, 2024, 45(11): 20240311.
ZHANG Lei, SHEN Huali. Quantitative Accuracy Evaluation of Mass Spectrometry Based Proteomics Methods Commonly Used in Biomarker Research. Chem. J. Chinese Universities, 2024, 45(11): 20240311.
| No. | Peptide sequence | No. | Peptide sequence |
|---|---|---|---|
| 1 | VSVHVIEGDHR | 17 | YLENYDAIR |
| 2 | AVTEQGHELSNEER | 18 | EANLLNAVIVQR |
| 3 | LVGSQEELASWGHEYVR | 19 | TEHINIHQLR |
| 4 | AQLFALTGVQPAR | 20 | AIGSASEGAQSSLQEVYHK |
| 5 | IQALQQQADEAEDR | 21 | TAFQEALDAAGDK |
| 6 | LVLVGDGGTGK | 22 | VYALPEDLVEVNPK |
| 7 | EAAENSLVAYK | 23 | NIETIINTFHQYSVK |
| 8 | SVTEQGAELSNEER | 24 | SGGASHSELIHNLR |
| 9 | FASENDLPEWK | 25 | SAGPQSQLLASVIAEK |
| 10 | LQDAEIAR | 26 | YFHVVIAGPQDSPFEGGTFK |
| 11 | LALQAQPVPDELVTK | 27 | TEGDGVYTLNNEK |
| 12 | FSGWYDADLSPAGHEEAK | 28 | VILENIASHEPR |
| 13 | IIFEDDR | 29 | ESSSHHPGIAEFPSR |
| 14 | AIVQLVNER | 30 | LVNEVTEFAK |
| 15 | VSEEIEDIIK | 31 | EQLTPLIK |
| 16 | HLFTGPVLSK | 32 | VGAHAGEYGAEALER |
Table 1 Detail of the 32 peptide sequence
| No. | Peptide sequence | No. | Peptide sequence |
|---|---|---|---|
| 1 | VSVHVIEGDHR | 17 | YLENYDAIR |
| 2 | AVTEQGHELSNEER | 18 | EANLLNAVIVQR |
| 3 | LVGSQEELASWGHEYVR | 19 | TEHINIHQLR |
| 4 | AQLFALTGVQPAR | 20 | AIGSASEGAQSSLQEVYHK |
| 5 | IQALQQQADEAEDR | 21 | TAFQEALDAAGDK |
| 6 | LVLVGDGGTGK | 22 | VYALPEDLVEVNPK |
| 7 | EAAENSLVAYK | 23 | NIETIINTFHQYSVK |
| 8 | SVTEQGAELSNEER | 24 | SGGASHSELIHNLR |
| 9 | FASENDLPEWK | 25 | SAGPQSQLLASVIAEK |
| 10 | LQDAEIAR | 26 | YFHVVIAGPQDSPFEGGTFK |
| 11 | LALQAQPVPDELVTK | 27 | TEGDGVYTLNNEK |
| 12 | FSGWYDADLSPAGHEEAK | 28 | VILENIASHEPR |
| 13 | IIFEDDR | 29 | ESSSHHPGIAEFPSR |
| 14 | AIVQLVNER | 30 | LVNEVTEFAK |
| 15 | VSEEIEDIIK | 31 | EQLTPLIK |
| 16 | HLFTGPVLSK | 32 | VGAHAGEYGAEALER |
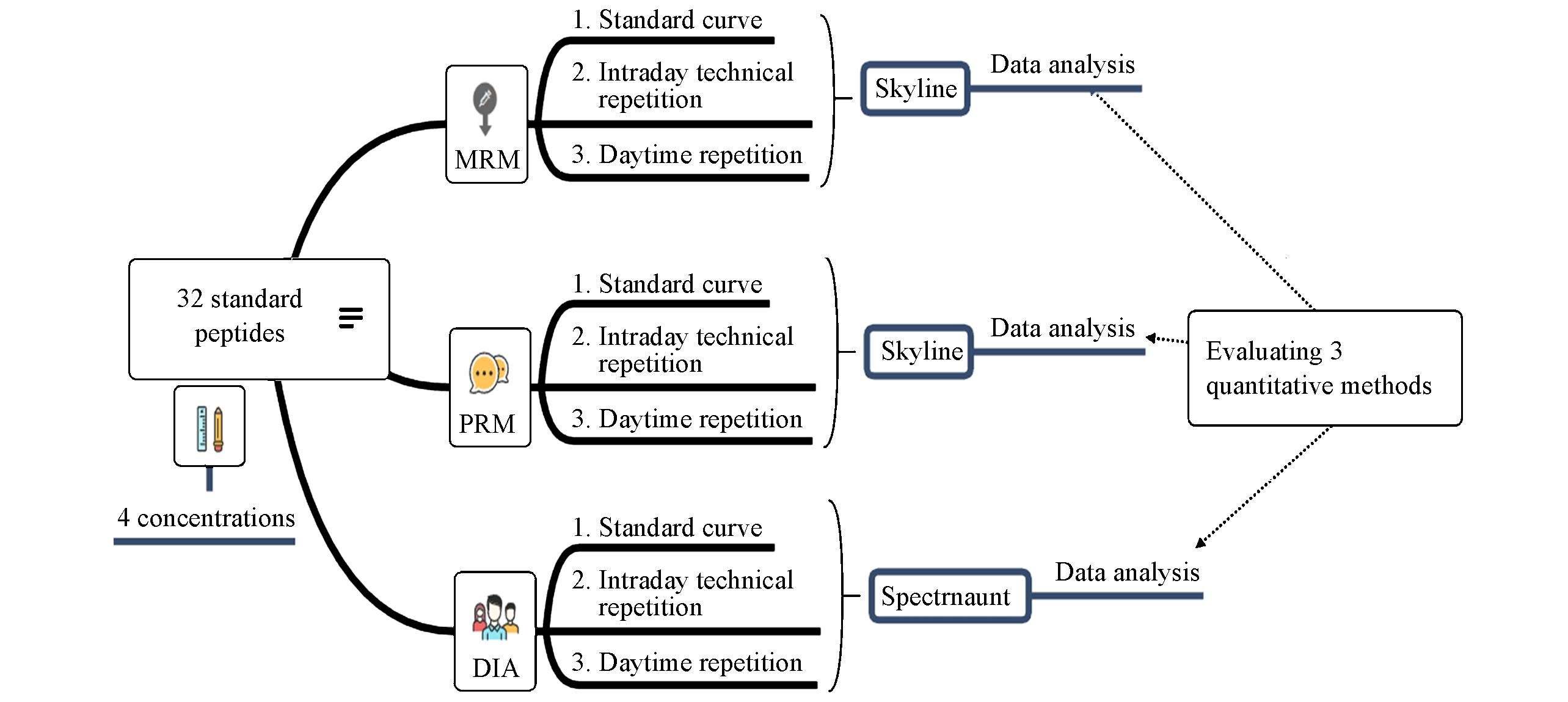
Fig.1 Evaluation of three quantitative methods using 32 standard peptides at four concentrationsEach method includes the creation of a standard curve, intraday technical repetition, and daytime repetition. Data analysis is performed using Skyline for MRM and PRM, and Spectronaut for DIA, to evaluate the performance and reliability of these quantitative methods.
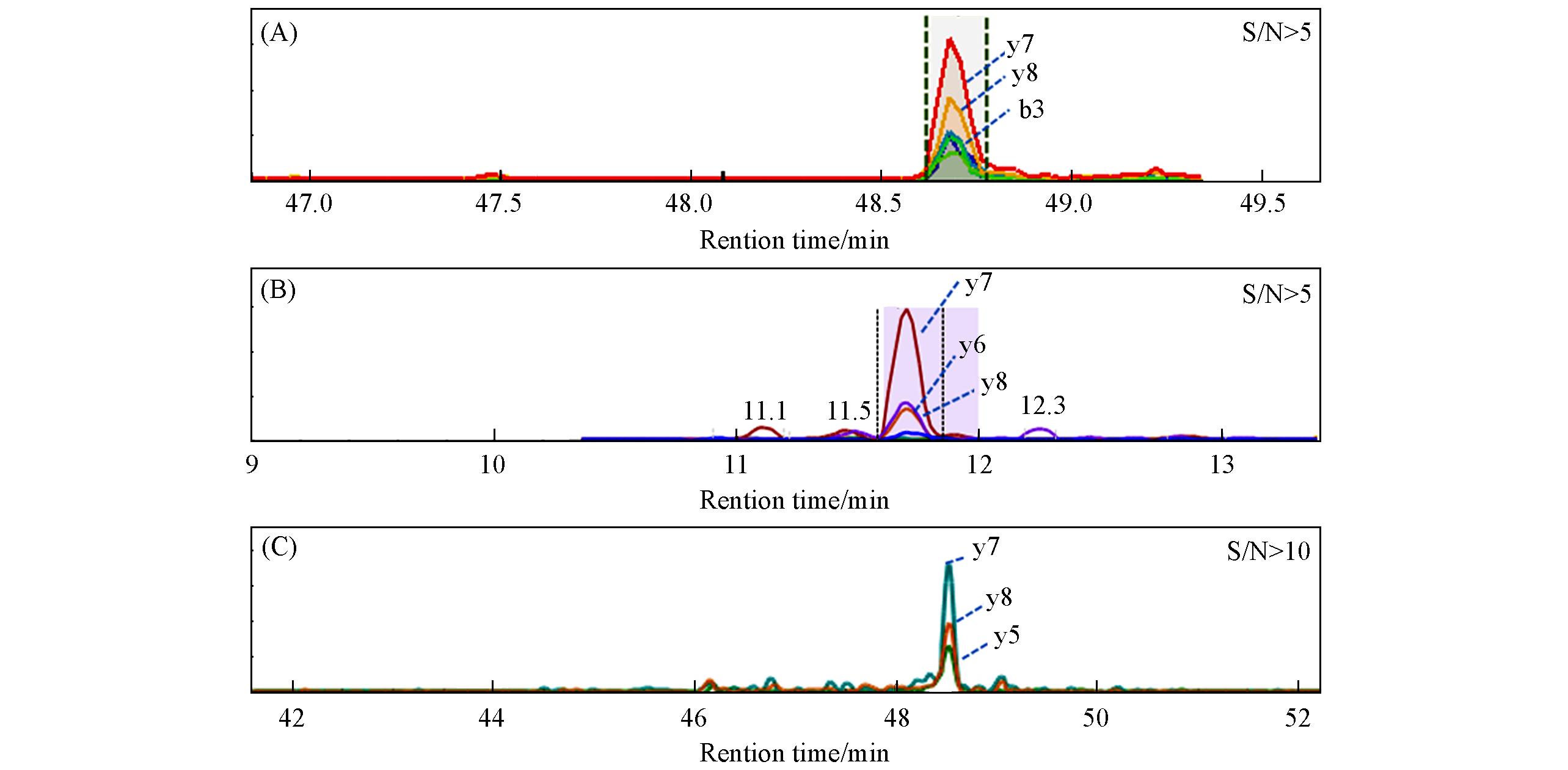
Fig.2 Detection and quantification of peptide EANLLNAVIVQR at 8 fmol/μL using DIA(A), MRM(B), and PRM(C) methodsDifferent colors represent different MS2 fragments.
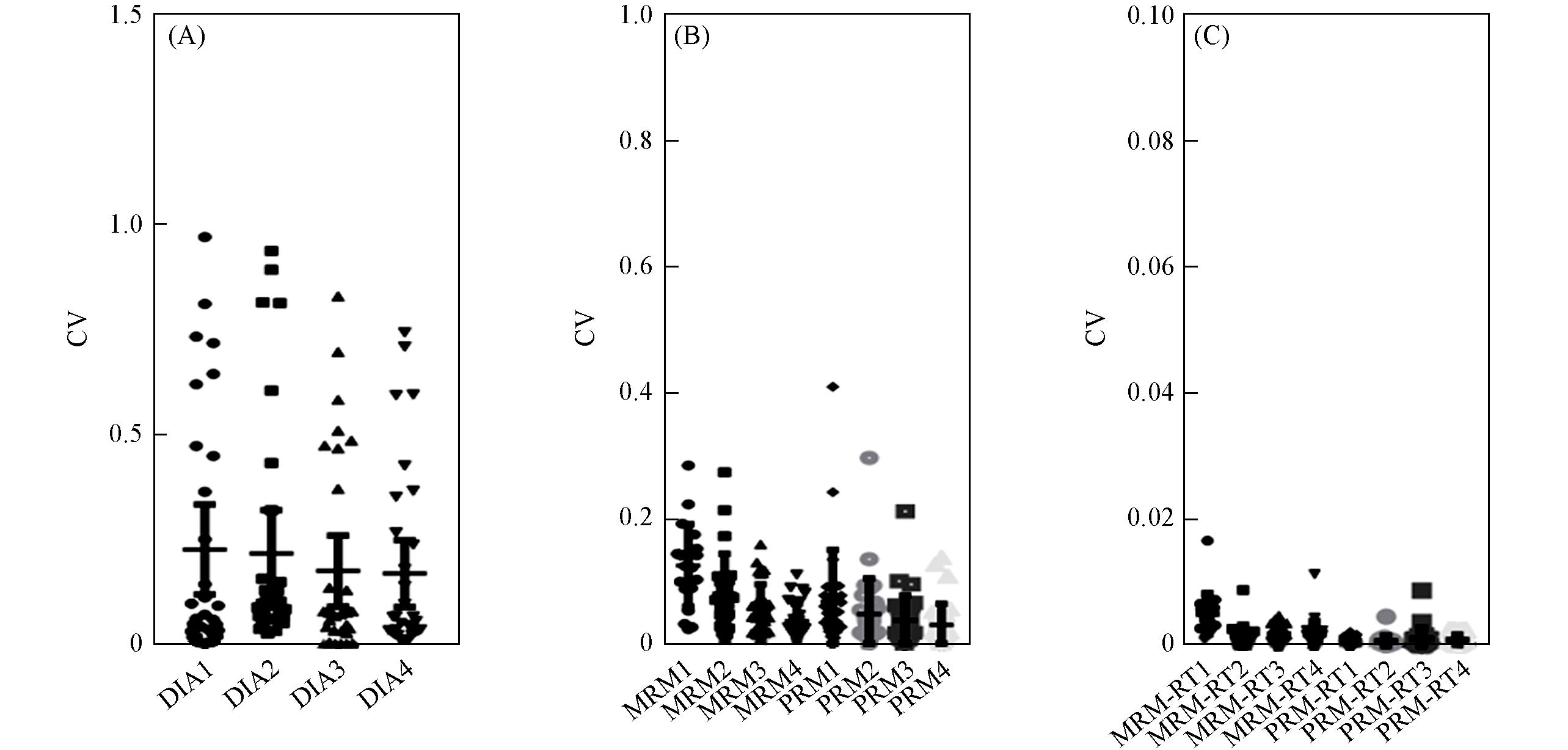
Fig.3 Coefficient of variation(CV) analysis for peptide quantification methods(A) Using DIA across four concentrations; (B) using MRM and PRM; (C) comparison of CV for the retention time(RT) between MRM and PRM across four replicates.
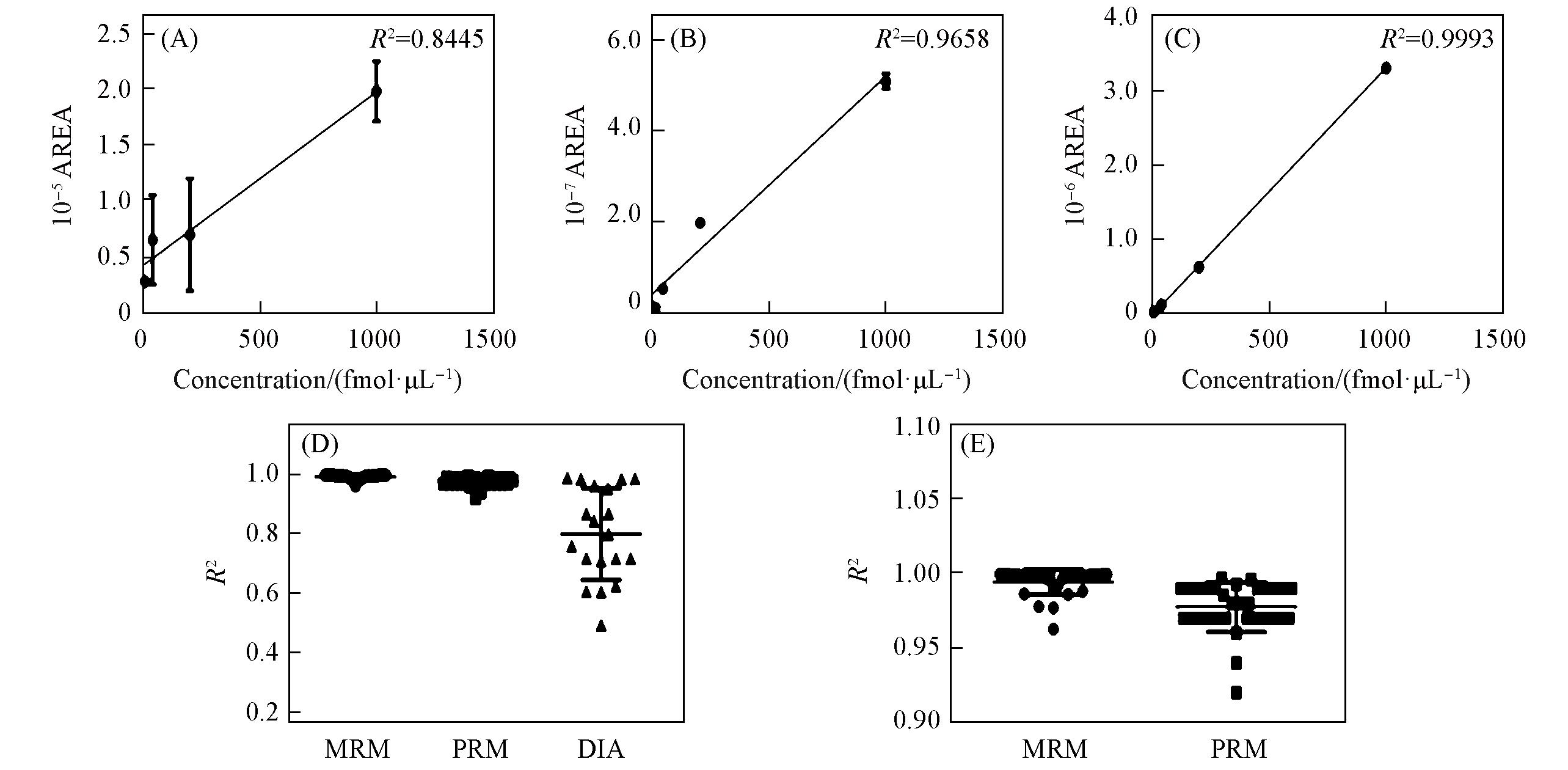
Fig.4 Quantitative performance using DIA, PRM, and MRM methods on peptide TEHINIHQLR(A—C) Standard curves using DIA, PRM, and MRM respectively; (D) comparison of R2 in the quantification of the 32 peptides across MRM, PRM, and DIA methods, indicating the linearity of the quantification for each method; (E) detailed comparison of R2 for MRM and PRM methods, showing high reproducibility and accuracy in the quantification.
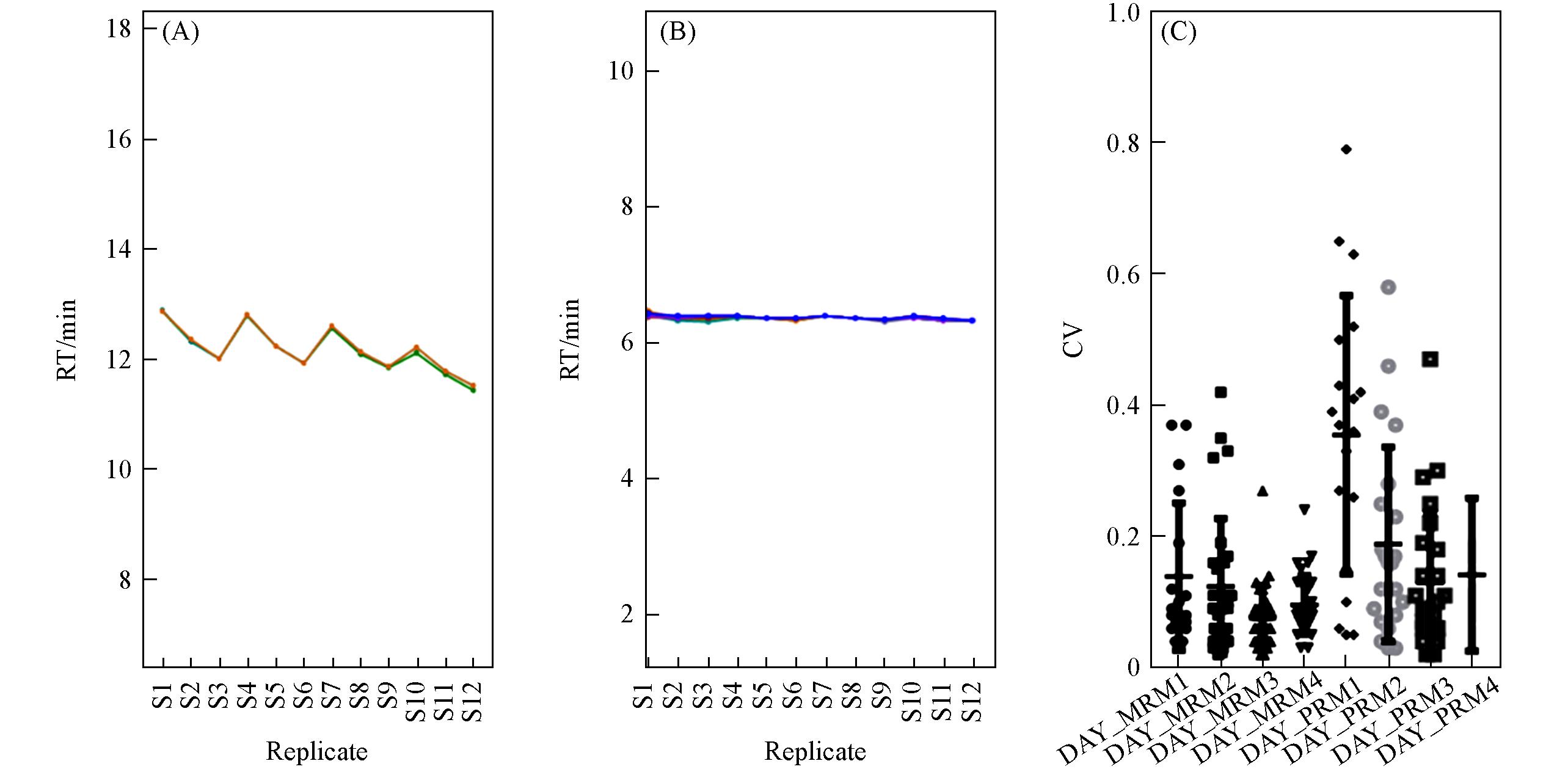
Fig.5 Daytime RT shift and CV of peak areas for PRM(A) and MRM(B) methods of VSVHVIEGDHR and CVs of peak areas between MRM and PRM methods across different days(C)
| 6 | Gao Q., Zhu H. W., Dong L. Q., Shi W. W., Chen R., Song Z. J., Huang C., Li J. Q., Dong X. W., Zhou Y. T., Liu Q., Ma L. J., Wang X. Y., Zhou J., Liu Y. S., Boja E., Robles A. I., Ma W. P., Wang P., Li Y. Z., Ding L., Wen B., Zhang B., Rodriguez H., Gao D. M., Zhou H., Fan J., Cell, 2019, 179(5), 561—577 |
| 7 | Tian W. M., Zhang N., Jin R. H., Feng Y. M., Wang S. Y., Gao S. X., Gao R. Q., Wu G. Z., Tian D., Tan W. J., Chen Y., Gao G. F., Wong C. C. L., Nature Commun., 2020,11(1), 5859 |
| 8 | Xu J. Y., Zhang C., Wang X., Zhai L., Ma Y., Mao Y., Qian K., Sun C., Liu Z., Jiang S., Wang M., Feng L., Zhao L., Liu P., Wang B., Zhao X., Xie H., Yang X., Zhao L., Chang Y., Jia J., Wang X., Zhang Y., Wang Y., Yang Y., Wu Z., Yang L., Liu B., Zhao T., Ren S., Sun A., Zhao Y., Ying W., Wang F., Wang G., Zhang Y., Cheng S., Qin J., Qian X., Wang Y., Li J., He F., Xiao T., Tan M., Cell, 2020, 182(1), 245—261 |
| 9 | Guo Y., You J., Zhang Y., Liu W. S., Huang Y. Y., Zhang Y. R., Zhang W., Dong Q., Feng J. F., Cheng W., Yu J. T., Nature Aging, 2024, 4(2), 247—260 |
| 10 | You J., Guo Y., Zhang Y., Kang J. J., Wang L. B., Feng J. F., Cheng W., Yu J. T., Nature Commun., 2023, 14(1), 7817 |
| 11 | Hoshino A., Kim H.S., Bojmar L., Gyan K. E., Cioffi M., Hernandez J., Zambirinis C. P., Rodrigues G., Molina H., Heissel S., Mark M. T., Steiner L., Benito⁃Martin A., Lucotti S., Di Giannatale A., Offer K., Nakajima M., Williams C., Nogués L., Vatter F. A. P., Hashimoto A., Davies A. E., Freitas D., Kenific C. M., Ararso Y., Buehring W., Lauritzen P., Ogitani Y., Sugiura K., Takahashi N., Aleckovic M., Bailey K. A., Jolissant J. S., Wang H. J., Harris A., Schaeffer L. M., García⁃Santos G., Posner Z., Balachandran V. P., Khakoo Y., Raju G. P., Scherz A., Sagi I., Scherz⁃Shouval R., Yarden Y., Oren M., Malladi M., Petriccione M., De Braganca K. C., Donzelli M., Fischer C., Vitolano S., Wright G. P., Ganshaw L., Marrano M., Ahmed A., DeStefano J., Danzer E., Roehrl M. H. A., Lacayo N. J., Vincent T. C., Weiser M. R., Brady M. S., Meyers P. A., Wexler L. H., Ambati S. R., Chou A. J., Slotkin E. K., Modak S., Roberts S. S., Basu E. M., Diolaiti D., Krantz B. A., Cardoso F., Simpson A. L., Berger M., Rudin C. M., Simeone D. M., Jain M., Ghajar C. M., Batra S. K., Stanger B., Bui J., Brown K. A., Rajasekhar V. K., Healey J. H., de Sousa M., Kramer K., Sheth S., Baisch J., Pascual V., Heaton T. E., La Quaglia M. P., Pisapia D. J., Schwartz R., Zhang H. Y., Liu Y., Shukla A., Blavier L., DeClerck Y. A., LaBarge M., Bissell M. J., Caffrey T. C., Grandgenett P. M., Hollingsworth M. A., Bromberg J., Costa⁃Silva B., Peinado H., Kang Y. B., Garcia B. A., O'Reilly E. M., Kelsen D., Trippett T. M., Jones D. R., Matei I. R., Jarnagin W. R., Lyden D., Cell, 2020, 182(4),1044—1061 |
| 12 | Shen B., Yi X., Sun Y. T., Bi X. J., Du J. P., Zhang C., Quan S., Zhang F. F., Sun R., Qian L. J., Ge W. G., Liu W., Liang S., Chen H., Zhang Y., Li J., Xu J. Q., He Z. B., Chen B. F., Wang J., Yan H. X., Zheng Y. F., Wang D. L., Zhu J. S., Kong Z. Q., Kang Z. Y., Liang X., Ding X., Ruan G., Xiang N., Cai X., Gao H. H., Li L., Li S. N., Xiao Q., Lu T., Zhu Y., Liu H. F., Chen H. X., Guo T. N., Cell, 2020, 182(1), 59—72 |
| 13 | Hu A. Q., Zhang L., Wang Z. X., Yuan C. Y., Lin L., Zhang J. Y., Gao X., Chen X. G., Guo W., Yang P. Y., Shen H. L., Anal. Chem., 2023, 95(2), 862—871 |
| 14 | Zhou M. T., Haque R. U., Dammer E. B., Duong D. M., Ping L.Y., Johnson E. C. B., Lah J. J., Levey A. I., Seyfried N. T., Clinical Proteomics, 2020, 17(1), 19 |
| 15 | Demichev V., Messner C. B., Vernardis S. I., Lilley K. S., Ralser M., Nature Methods, 2020, 17(1), 41—44 |
| 16 | Messner C. B., Demichev V., Bloomfield N., Yu J. S. L., White M., Kreidl M., Egger A. S., Freiwald A., Ivosev G., Wasim F., Zelezniak A., Jürgens L., Suttorp N., Sander L. E., Kurth F., Lilley K. S., Mülleder M., Tate S., Ralser M., Nature Biotechnology, 2021, 39(7), 846—854 |
| 17 | Vidova V., Spacil Z., Anal. Chim. Acta, 2017, 964, 7—23 |
| 18 | Zecha J., Lee C. Y., Bayer F. P., Meng C., Vincent, Zerweck J., Schnatbaum K., Michler T., Pichlmair A., Ludwig C., Kuster B., Molecular & Cellular Proteomics, 2020, 19(9), 1503—1521 |
| 1 | Kim M. S., Pinto S. M., Getnet D., Nirujogi R. S., Manda S. S., Chaerkady R., Madugundu A. K., Kelkar D. S., Isserlin R., Jain S., Thomas J. K., Muthusamy B., Leal⁃Rojas P., Kumar P., Sahasrabuddhe N.A., Balakrishnan L., Advani J., George B., Renuse S., Selvan L. D. N., Patil A. H., Nanjappa V., Radhakrishnan A., Prasad S., Subbannayya T., Raju R., Kumar M., Sreenivasamurthy S. K., Marimuthu A., Sathe G. J., Chavan S., Datta K. K., Subbannayya Y., Sahu A., Yelamanchi S.D., Jayaram S., Rajagopalan P., Sharma J., Murthy K. R., Syed N., Goel R., Khan A. A., Ahmad S., Dey G., Mudgal K., Chatterjee A., Huang T. C., Zhong J., Wu X. Y., Shaw P. G., Freed D., Zahari M. S., Mukherjee K. K., Shankar S., Mahadevan A., Lam H., Mitchell C. J., Shankar S. K., Satishchandra P., Schroeder J.T., Sirdeshmukh R., Maitra A., Leach S.D., Drake C.G., Halushka M. K., Prasad T. S. K., Hruban R. H., Kerr C. L., Bader G. D., Iacobuzio⁃Donahue C. A., Gowda H., Pandey A., Nature, 2014, 509(7502), 575—581 |
| 2 | Wilhelm M., Schlegl J., Hahne H., Gholami A. M., Lieberenz M., Savitski M. M., Ziegler E., Butzmann L., Gessulat S., Marx H., Mathieson T., Lemeer S., Schnatbaum K., Reimer U., Wenschuh H., Mollenhauer M., Slotta-Huspenina J., Boese J. H., Bantscheff M., Gerstmair A., Faerber F., Kuster B., Nature, 2014, 509(7502), 582—587 |
| 3 | Uhlen M., Fagerberg L., Hallström B.M., Lindskog C., Oksvold P., Mardinoglu A., Sivertsson A., Kampf C., Sjöstedt E., Asplund A., Olsson I., Edlund K., Lundberg E., Navani S., Szigyarto C. A., Odeberg J., Djureinovic D., Takanen J.O., Hober S., Alm T., Edqvist P.H., Berling H., Tegel H., Mulder J., Rockberg J., Nilsson P., Schwenk J. M., Hamsten M., von Feilitzen K., Forsberg M., Persson L., Johansson F., Zwahlen M., von Heijne G., Nielsen J., Pontén F., Science, 2015, 347(6220), 1260419 |
| 4 | Zhang H., Liu T., Zhang Z., Payne S.H., Zhang B., McDermott J. E., Zhou J. Y., Petyuk V. A., Chen L., Ray D., Sun S. S., Yang F., Chen L. J., Wang J., Shah P., Cha S. W., Aiyetan P., Woo S., Tian Y., Gritsenko M. A., Clauss T. R., Choi C., Monroe M. E., Thomas S., Nie S., Wu C. C., Moore R. J., Yu K. H., Tabb D. L., Fenyö D., Bafna V., Wang Y., Rodriguez H., Boja E. S., Hiltke T., Rivers R. C., Sokoll L., Zhu H., Shih I. M., Cope L., Pandey A., Zhang B., Snyder M. P., Levine D. A., Smith R. D., Chan D. W., Rodland K. D., Investigators C., Cell, 2016, 166(3), 755—765 |
| 5 | Jiang Y., Sun A. H., Zhao Y., Ying W. T., Sun H. C., Yang X. R., Xing B. C., Sun W., Ren L. L., Hu B., Li C. Y., Zhang L., Qin G. R., Zhang M. H., Chen N., Zhang M. L., Huang Y., Zhou J. N., Zhao Y., Liu M. W., Zhu X. D., Qiu Y., Sun Y. J., Huang C., Yan M., Wang M. C., Liu W., Tian F., Xu H. L., Zhou J., Wu Z. Y., Shi T. L., Zhu W. M., Qin J., Xie L., Fan J., Qian X. H., He F. C., CNHPP Consortium, Nature, 2019, 567(7744), 257—261 |
| 19 | Heil L. R., Damoc E., Arrey T. N., Pashkova A., Denisov E., Petzoldt J., Peterson A. C., Hsu C., Searle B. C., Shulman N., Riffle M., Connolly B., MacLean B. X., Remes P. M., Senko M. W., Stewart H. I., Hock C., Makarov A. A., Hermanson D., Zabrouskov V., Wu C. C., MacCoss M. J., J. Proteome Res., 2023, 22(10), 3290—3300 |
| 20 | Niu L., Geyer P. E., Albrechtsen N. J. W., Gluud L. L., Santos A., Doll S., Treit P. V., Holst J. J., Knop F. K., Vilsbol T., Junker A., Sachs S., Stemmer K., Müller T. D., Tschöp M. H., Hofmann S. M., Mann M., Molecular Systems Biology, 2019, 15(3), e8793 |
| 21 | Niu L. L., Thiele M., Geyer P., Rasmussen D. N., Webel H. E., Delgado A., Gupta R., Meier F., Strauss M., Kjærgaard M., Lindvig K. P., Israelsen M., Rasmussen S., Hansen T., Mann M., Krag A., J. Hepatology, 2022, 77, 500—501 |
| 22 | Niu L. L., Thiele M., Geyer P. E., Rasmussen D. N., Webel H. E., Santos A., Gupta R., Meier F., Strauss M., Kjaergaard M., Lindvig K., Jacobsen S., Rasmussen S., Hansen T., Krag A., Mann M., Nature Medicine, 2022, 28(6), 1277—1287 |
| 23 | Grossegesse M., Hartkopf F., Nitsche A., Doellinger J., J. Proteome Research, 2020, 19(7), 2854—2862 |
| 24 | Zhu H., Ficarro S. B., Alexander W. M., Fleming L. E., Adelmant G., Zhang T. H., Willetts M., Decker J., Brehmer S., Krause M., East M. P., Gray N. S., Johnson G. L., Kruppa G., Marto J. A., Anal. Chem., 2021, 93(41), 13791—13799 |
| 25 | Wu W., Zhou Q. P., Masubuchi T., Shi X. S., Li H., Xu X. Y., Huang M., Meng L., He X., Zhu H. Y., Gao S. X., Zhang N., Jing R. R., Sun J., Wang H. P., Hui E. F., Wong C. C., Xu C. Q., Cell, 2020, 182(4), 855—871 |
| 26 | Wang Z. X., Wang H., Peng Y. F., Chen F. J., Zhao L., Li X. M., Qin J. Q., Li Q. Q., Wang B. L., Pan B. S., Guo W., Clinical Chemistry and Laboratory Medicine, 2020, 58(9), 1477—1487 |
| [1] | 靳莹, 张俊杰, 张毅欣, 袁悦, 韩珍珍. 外泌体分离和蛋白质组学分析的研究进展[J]. 高等学校化学学报, 2024, 45(11): 20240305. |
| [2] | 胡宇虹, 俞相明, 宋丽丽, 邢清和, 周峰. DEEP SEQ方法检测新生儿毛干蛋白质组动态变化[J]. 高等学校化学学报, 2024, 45(11): 20240326. |
| [3] | 范智瑞, 方群, 杨奕. 基于质谱的单细胞蛋白质组学分析[J]. 高等学校化学学报, 2024, 45(11): 20240294. |
| [4] | 许霞, 秦伟达, 李若萌, 王倩倩, 刘宁, 李功玉. 深度覆盖蛋白质组学质谱分析: 细胞蛋白提取方法的评估[J]. 高等学校化学学报, 2024, 45(11): 20240344. |
| [5] | 黄玉滢, 于成鲲, 刘斯奇, 任艳. 利用无标记单细胞蛋白质组学方法构建小鼠外周血单个核细胞的细胞图谱[J]. 高等学校化学学报, 2024, 45(11): 20240355. |
| [6] | 沈枫林, 冯兆莹, 方静, 张磊, 刘晓慧, 周新文. 基于质谱的单细胞分辨的空间蛋白质组学新技术研究[J]. 高等学校化学学报, 2024, 45(11): 20240299. |
| [7] | 霍志远, 周金萍, 马秀敏, 周严, 黄琳. 基于质谱的单细胞多组学分析技术研究进展[J]. 高等学校化学学报, 2024, 45(11): 20240389. |
| [8] | 董沛滢, 刘彤, 秦伟捷. RNA-蛋白质复合物规模化富集与鉴定新方法[J]. 高等学校化学学报, 2024, 45(11): 20240091. |
| [9] | 赵欢欢, 李存玉, 金红. 基于TMT10-plex等量标记结合SMOAC富集磷酸肽的定量磷酸化蛋白质组性能评价[J]. 高等学校化学学报, 2021, 42(12): 3624. |
| [10] | 魏王慧, 储艳秋, 陈鹰, 高艳秋, 丁传凡. 质谱法定量测定高密度脂蛋白结合蛋白的糖基化水平[J]. 高等学校化学学报, 2019, 40(6): 1141. |
| [11] | 李颖, 姚鋆, 杨芃原, 樊惠芝. 液相等电聚焦技术结合液相色谱-质谱联用技术分析鼠肝全蛋白[J]. 高等学校化学学报, 2015, 36(6): 1074. |
| [12] | 郑君芳, 华琳, 张秋霞, 李德龙, 贺俊崎. 嗅觉记忆相关蛋白的蛋白质组学研究[J]. 高等学校化学学报, 2010, 31(8): 1535. |
| [13] | 郑君芳, 刘华, 熊英, 王小柱, 贺俊崎. 恐惧记忆相关蛋白的蛋白质组学研究[J]. 高等学校化学学报, 2010, 31(4): 736. |
| [14] | 方财王, 黄清育, 凌雪萍, 柯才焕, 黄河清. 在镉盐胁迫下扇贝鳃组织应激蛋白的研究[J]. 高等学校化学学报, 2010, 31(3): 507. |
| [15] | 赵慧辉, 杨帆, 王伟, 王红霞, 魏开华. 无标记定量法研究冠心病不稳定性心绞痛血瘀证的差异蛋白质组[J]. 高等学校化学学报, 2010, 31(2): 285. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
