

高等学校化学学报 ›› 2024, Vol. 45 ›› Issue (7): 20240139.doi: 10.7503/cjcu20240139
• 综合评述 • 上一篇
肖航1,2, 王小燕1,2, 邓兆佳1,2, 廖文静1,2,3, 谢文菁1,2, 彭汉勇1,2( )
)
收稿日期:2024-03-25
出版日期:2024-07-10
发布日期:2024-05-15
通讯作者:
彭汉勇
E-mail:hypeng@rcees.ac.cn
作者简介:第一联系人:共同第一作者.
基金资助:
XIAO Hang1,2, WANG Xiaoyan1,2, DENG Zhaojia1,2, LIAO Wenjing1,2,3, XIE Wenjing1,2, PENG Hanyong1,2( )
)
Received:2024-03-25
Online:2024-07-10
Published:2024-05-15
Contact:
PENG Hanyong
E-mail:hypeng@rcees.ac.cn
Supported by:摘要:
病毒是引发人类疾病的主要病原体之一. 传统的聚合酶链式反应(PCR)技术虽然被广泛应用于病毒分子诊断, 但其对温度的要求较为严格, 限制了其在现场诊断中的应用. 为了满足现场快速诊断的需求, 核酸等温扩增技术得到快速发展, 其无需热循环, 可以在恒定温度下实现核酸扩增, 可适应不同的应用场景. 本文综合评述了等温扩增技术在病毒检测领域的最新进展, 从病毒样本采集、 核酸提取、 等温扩增检测等几个方面分别进行阐述, 探讨了酶辅助等温扩增技术、 无酶等温扩增技术以及与多体系串联的级联扩增技术的原理、 关键参数及其病毒检测应用, 并对比了市场上相关试剂盒的特点. 此外, 讨论了当前核酸等温扩增技术在病原体检测应用中面临的一些难题, 如提取效率、 稳定性和成本等, 提出了未来的发展方向, 为进一步改善现场诊断效率提供了新的思路.
中图分类号:
TrendMD:
肖航, 王小燕, 邓兆佳, 廖文静, 谢文菁, 彭汉勇. 核酸等温扩增技术在病毒检测中的应用. 高等学校化学学报, 2024, 45(7): 20240139.
XIAO Hang, WANG Xiaoyan, DENG Zhaojia, LIAO Wenjing, XIE Wenjing, PENG Hanyong. Development of Nucleic Acid Isothermal Amplification Technologies for Virus Detection. Chem. J. Chinese Universities, 2024, 45(7): 20240139.
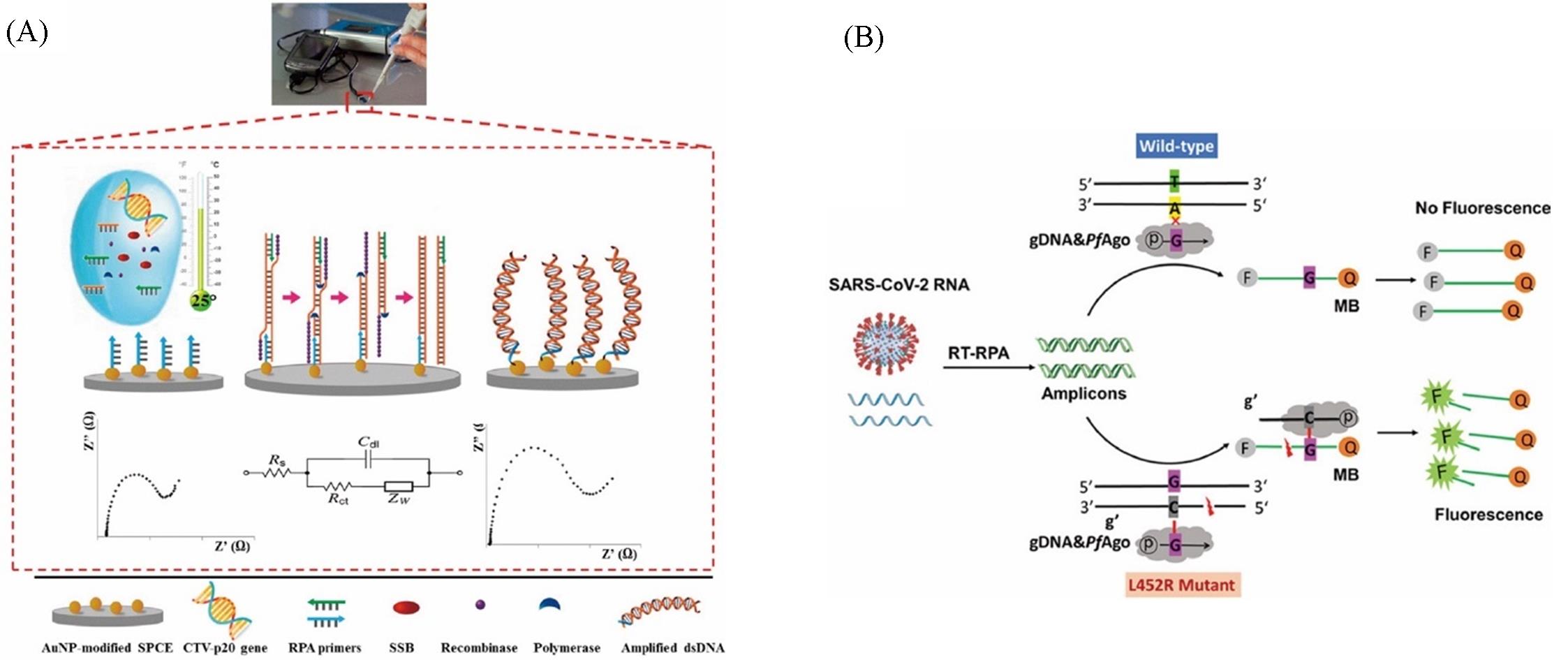
Fig.2 RPA⁃based virus detection method(A) Label⁃free in situ isothermal RPA amplification in electrochemical sensors[67]. Copyright 2019, American Chemical Society; (B) fluorescence detection of RT⁃RPA incorporated PfAgo enzyme[68]. Copyright 2022, American Chemical Society.
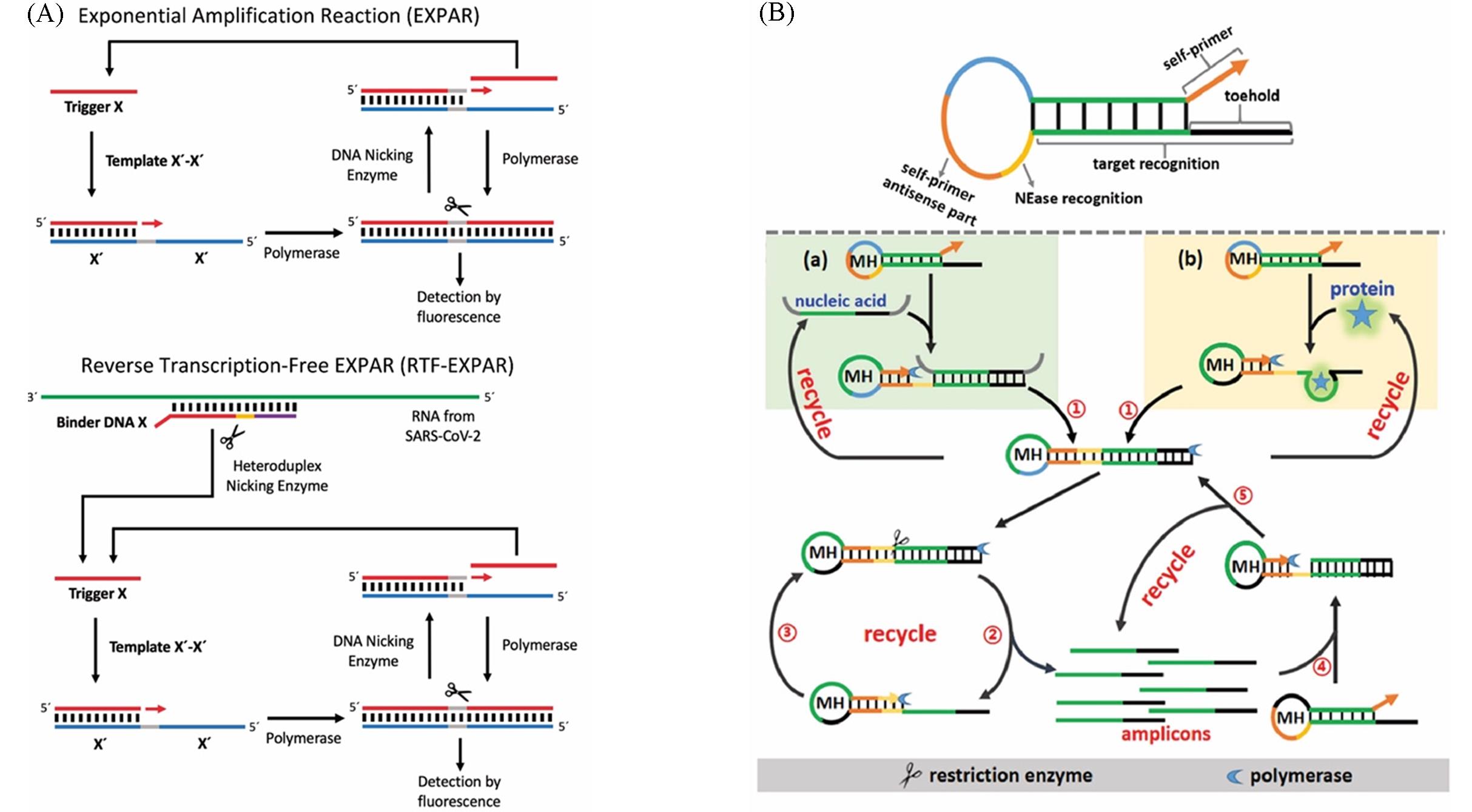
Fig.3 EXPAR⁃based virus detection method(A) Reverse transcription-free EXPAR[70]. Copyright 2021, National Academy of Sciences; (B)Self-prime EXPAR[71]. Copyright 2021, American Chemical Society.
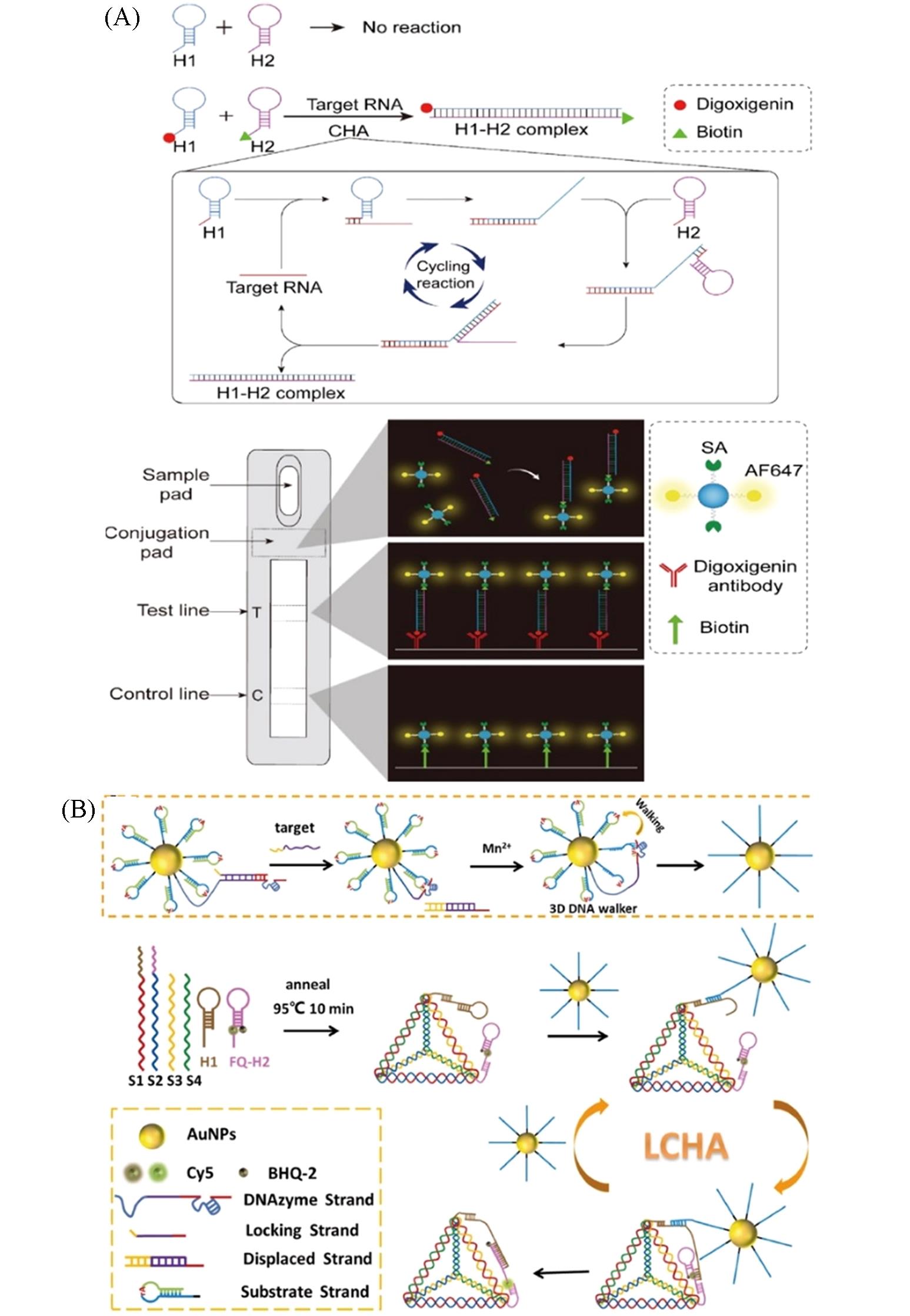
Fig.4 CHA⁃based virus detection method(A) CHA-Lateral flow immunoassay[87]. Copyright 2021, Elsevier; (B) LCHA-DNA walker fluorescence biosensor[88]. Copyright 2022, Elsevier.
| Method | Enzymes | Temperature/℃ | Reaction time/h | Efficiency | Ref. |
|---|---|---|---|---|---|
| NASBA | 2 | ca. 41 | 1.5—2 | 106—109 | [ |
| RCA | 2 | 60—65 | 1—3 | 103—109 | [ |
| SDA | 2 | 37—70 | 2 | 107 | [ |
| LAMP | 1 | 60—65 | 1—2 | 107—109 | [ |
| HDA | 2 | 37—65 | 0.5—2 | 106 | [ |
| RPA | 3 | 37—42 | 0.5—1.5 | 107—109 | [ |
| EXPAR | 2 | 60 | <0.5 | 106—108 | [ |
| CHA | — | 25—37 | 1 | 7000(two layers) | [ |
| HCR | — | 25—37 | — | — | [ |
| EDC | — | 25—37 | 1 | — | [ |
Table 1 Summary of isothermal amplification methods
| Method | Enzymes | Temperature/℃ | Reaction time/h | Efficiency | Ref. |
|---|---|---|---|---|---|
| NASBA | 2 | ca. 41 | 1.5—2 | 106—109 | [ |
| RCA | 2 | 60—65 | 1—3 | 103—109 | [ |
| SDA | 2 | 37—70 | 2 | 107 | [ |
| LAMP | 1 | 60—65 | 1—2 | 107—109 | [ |
| HDA | 2 | 37—65 | 0.5—2 | 106 | [ |
| RPA | 3 | 37—42 | 0.5—1.5 | 107—109 | [ |
| EXPAR | 2 | 60 | <0.5 | 106—108 | [ |
| CHA | — | 25—37 | 1 | 7000(two layers) | [ |
| HCR | — | 25—37 | — | — | [ |
| EDC | — | 25—37 | 1 | — | [ |
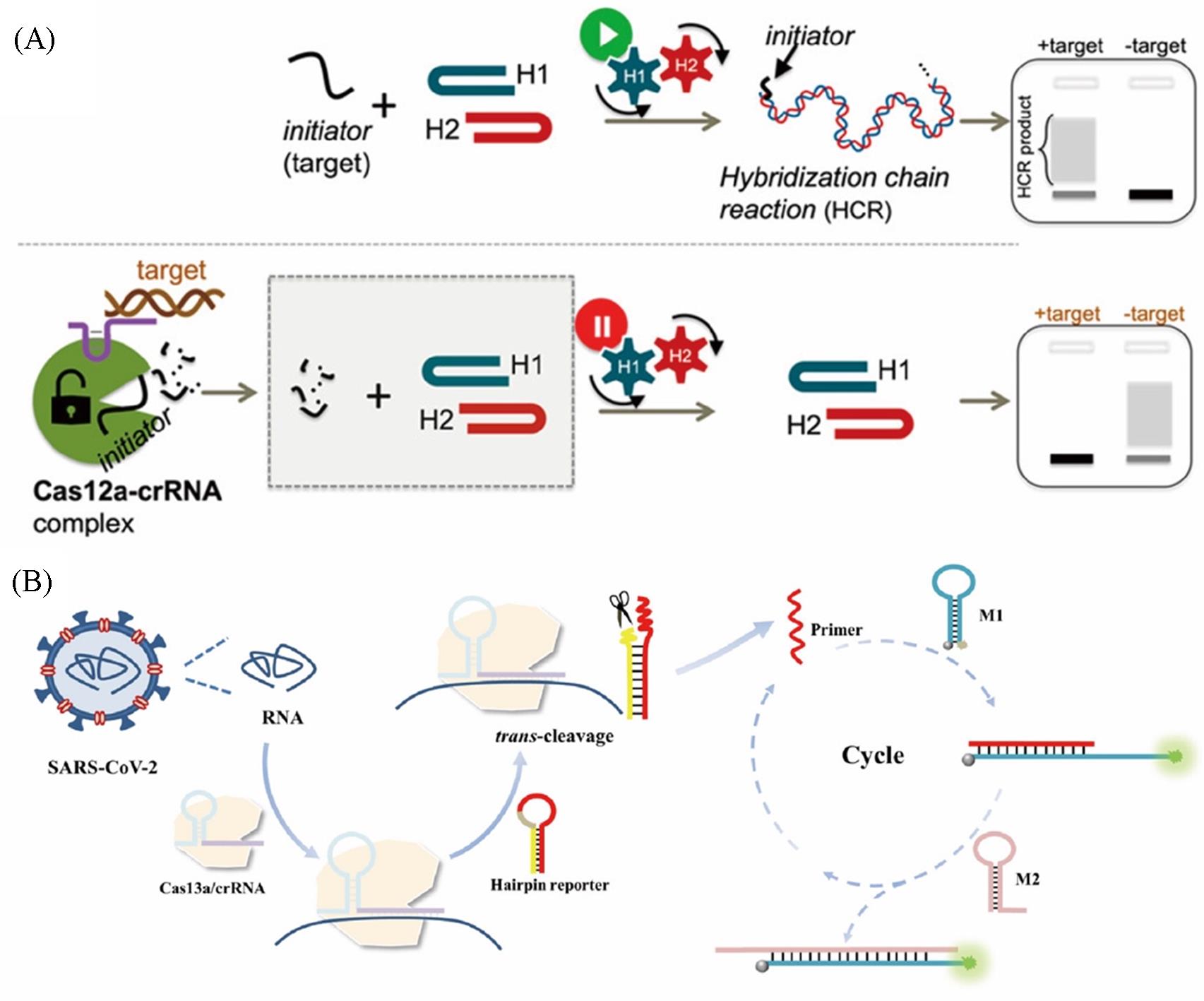
Fig.6 Enzyme⁃free nucleic acid isothermal amplification and CRISPR⁃Cas cascades(A) HCR and CRISPR-Cas12a system for TCSV and HepBV detection[104]. Copyright 2021, American Chemical Society; (B) CHA-Cas13a assay for SARS-CoV-2 detection[106]. Copyright 2023, American Chemical Society.
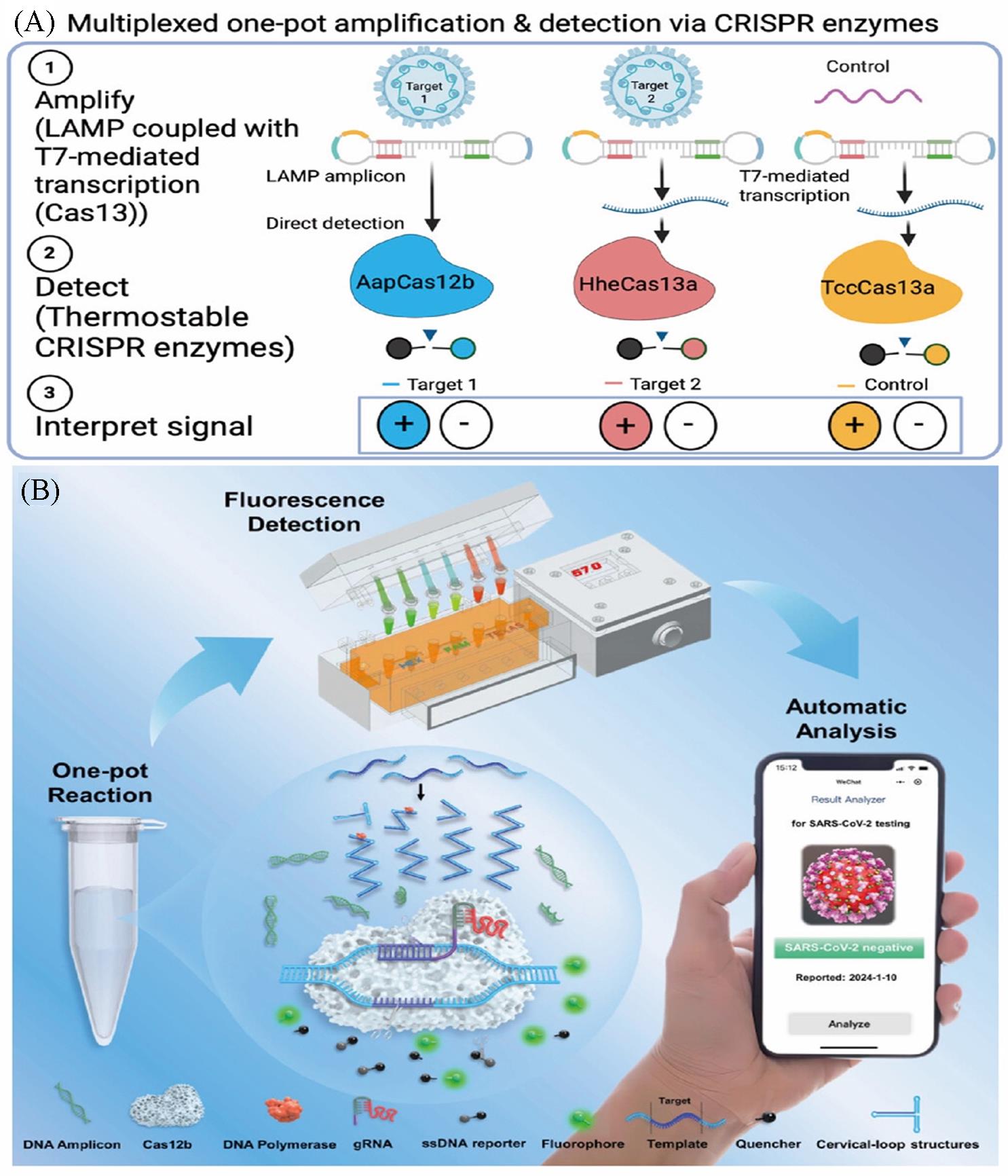
Fig.7 RPA and CRISPR⁃Cas based multiplex detection of viruses(A) CRISPR⁃Cas multiplexed diagnostic assay for hrHPV detection [110]. Copyright 2024, American Chemical Society; (B) one⁃pot CRISPR⁃based “green⁃yellow⁃red” multiplex detection CHA⁃Cas13a assay for SARS⁃CoV⁃2 detection [111]. Copyright 2024, American Chemical Society.
| Product | Technique | Target | Cost | Testing time | Type | Biotech company | Ref. |
|---|---|---|---|---|---|---|---|
| NucliSens EasyQ | NASBA | virus RNA | $50, 000 | 1 h | Automated analysis methods | Biomeriux | [ |
| OligoC⁃TesT | NASBA | Leishmania parasites DNA | $26/test | 1 h | Kit | Coris Bio Concept, Belguim | [ |
| BESt Cassette Type II | HDA | Staphylococcus aureus DNA | $10/test | 1 h | Kit | BioHelix, USA | [ |
| Illumigene(Alethia) | LAMP | Clostridium difficile DNA | $30.6/test | 40 min | Kit | Meridian Bioscience, USA | [ |
Loopamp MTBC Detection Kit® | LAMP | Mycobacterium tuberculosis DNA | $5.7/test | 30 min | Kit | Eiken Chemical Co, Japan | [ |
| Eazyplex® | LAMP | Bacterial pathogens | — | 30 min | Kit | Amplex Diagnostics GmbH, Germany | [ |
3MTM Molecular Detection System | LAMP | Salmonella DNA | — | 75 min | Kit | 3M, USA | [ |
| Genie III | LAMP | Ebola virus RNA | $13, 000 | 15-20 min | Automated analysis methods | OptiGene, UK | [ |
| Solana® | HDA | Trichomonas DNA | — | 1 h | Kit | Quidel Corporation, USA | [ |
| BD ProbeTecTM | SDA | Chlamydia trachomatis, gonorrhoeae DNA | — | 3 h | Kit/Automated analysis methods | BD Diagnostics, USA | [ |
Table 2 Summary of reagent kits and assay methods based on isothermal amplification technology
| Product | Technique | Target | Cost | Testing time | Type | Biotech company | Ref. |
|---|---|---|---|---|---|---|---|
| NucliSens EasyQ | NASBA | virus RNA | $50, 000 | 1 h | Automated analysis methods | Biomeriux | [ |
| OligoC⁃TesT | NASBA | Leishmania parasites DNA | $26/test | 1 h | Kit | Coris Bio Concept, Belguim | [ |
| BESt Cassette Type II | HDA | Staphylococcus aureus DNA | $10/test | 1 h | Kit | BioHelix, USA | [ |
| Illumigene(Alethia) | LAMP | Clostridium difficile DNA | $30.6/test | 40 min | Kit | Meridian Bioscience, USA | [ |
Loopamp MTBC Detection Kit® | LAMP | Mycobacterium tuberculosis DNA | $5.7/test | 30 min | Kit | Eiken Chemical Co, Japan | [ |
| Eazyplex® | LAMP | Bacterial pathogens | — | 30 min | Kit | Amplex Diagnostics GmbH, Germany | [ |
3MTM Molecular Detection System | LAMP | Salmonella DNA | — | 75 min | Kit | 3M, USA | [ |
| Genie III | LAMP | Ebola virus RNA | $13, 000 | 15-20 min | Automated analysis methods | OptiGene, UK | [ |
| Solana® | HDA | Trichomonas DNA | — | 1 h | Kit | Quidel Corporation, USA | [ |
| BD ProbeTecTM | SDA | Chlamydia trachomatis, gonorrhoeae DNA | — | 3 h | Kit/Automated analysis methods | BD Diagnostics, USA | [ |
| 1 | Zhu N., Zhang D., Wang W., Li X., Yang B., Song J., Zhao X., Huang B., Shi W., Lu R., N. Engl. J. Med., 2020, 382(8), 727—733 |
| 2 | Liu R., Han H., Liu F., Lv Z., Wu K., Liu Y., Feng Y., Zhu C., Clin. Chim. Acta, 2020, 505, 172—175 |
| 3 | Wu F., Zhao S., Yu B., Chen Y. M., Wang W., Song Z. G., Hu Y., Tao Z. W., Tian J. H., Pei Y. Y., Nature, 2020, 579(7798), 265—269 |
| 4 | Zhou P., Yang X. L., Wang X. G., Hu B., Zhang L., Zhang W., Si H. R., Zhu Y., Li B., Huang C. L., Nature, 2020, 579(7798), 270—273 |
| 5 | Corman V. M., Landt O., Kaiser M., Molenkamp R., Meijer A., Chu D. K., Bleicker T., Brünink S., Schneider J., Schmidt M. L., Eurosurveillance, 2020, 25(3), 2000045 |
| 6 | Zhao Y., Chen F., Li Q., Wang L., Fan C., Chem. Rev., 2015, 115(22), 12491—12545 |
| 7 | Wang W., Xu Y., Gao R., Lu R., Han K., Wu G., Tan W., J. Am. Med. Assoc., 2020, 323(18), 1843—1844 |
| 8 | Organization W. H., World Health Organization, 2020 |
| 9 | Henningson E. W., Ahlberg M. S., J. Aerosol. Sci., 1994, 25(8), 1459—1492 |
| 10 | Truyols V. J., Stiliyanov A. K., Sala L. E., Toledo P. N., Baldovi H. G., Mercader B. J., J. Hosp. Infect., 2022, 130, 44—51 |
| 11 | Tedeschini E., Pasqualini S., Emiliani C., Marini E., Valecchi A., Laoreti C., Ministrini S., Camilloni B., Castronari R., Patoia L., Merante F., Baglioni S., De R. E., Pirro M., Mencacci A., Pasqualini L., Front. Public. Health., 2023, 11, 1169073 |
| 12 | Tan K. S., Ang A. X. Y., Tay D. J. W., Somani J., Ng A. J. Y., Peng L. L., Chu J. J. H., Tambyah P. A., Allen D. M., Front. Public. Health., 2023, 10, 1067575 |
| 13 | Qiu G., Spillmann M., Tang J., Zhao Y. B., Tao Y., Zhang X., Geschwindner H., Saleh L., Zingg W., Wang J., Adv. Sci, 2022, 9(35), 2204774 |
| 14 | Alonso C., Raynor P. C., Davies P. R., Torremorell M., PLoS One, 2015, 10(8), e0135675 |
| 15 | Silva P. G., Branco P. T. B. S., Soares R. R. G., Mesquita J. R., Sousa S. I. V., Indoor Air, 2022, 32(8), e13083 |
| 16 | Verreault D., Moineau S., Duchaine C., Microbiol. Mol. Biol. Rev., 2008, 72(3), 413—444 |
| 17 | Bazzazpour S., Rahmatinia M., Mohebbi S. R., Hadei M., Shahsavani A., Hopke P. K., Houshmand B., Raeisi A., Jafari A. J., Yarahmadi M., Farhadi M., Hasanzadeh V., Kermani M., Vaziri M. H., Tanhaei M., Zali M. R., Alipour M. R., Environ. Sci. Pollut. Res., 2022, 29(57), 85586—85594 |
| 18 | Feng X. L., Li B., Lin H. F., Zheng H. Y., Tian R. R., Luo R. H., Liu M. Q., Jiang R. D., Zheng Y. T., Shi Z. L., Bi Y. H., Yang X. L., Virol. Sin., 2021, 36(5), 1069—1072 |
| 19 | Park G. W., Lee D., Treffiletti A., Hrsak M., Shugart J., Vinje J., Appl. Environ. Microbiol., 2015, 81(17), 5987—5992 |
| 20 | Kong J., Li W., Hu J., Zhao S., Yue T., Li Z., Xia Y., Foods, 2022, 11(11), 1540 |
| 21 | Santiago R. T. M., Water, 2022, 14(4), 599 |
| 22 | Ahmed W., Bivins A., Bertsch P. M., Bibby K., Choi P. M., Farkas K., Gyawali P., Hamilton K. A., Haramoto E., Kitajima M., Simpson S. L., Tandukar S., Thomas K. V., Mueller J. F., Curr. Opin. Environ. Sci. Health, 2020, 17, 82—93 |
| 23 | Buonerba A., Corpuz M. V. A., Ballesteros F., Choo K. H., Hasan S. W., Korshin G. V., Belgiorno V., Barcelo D., Naddeo V., J. Hazard. Mater., 2021, 415, 125580 |
| 24 | Cervantes Aviles. P., Moreno Andrade. I., Carrillo Reyes. J., J. Water Process. Eng., 2021, 40, 101947 |
| 25 | Sim S. L., He T., Tscheliessnig A., Mueller M., Tan R. B. H., Jungbauer A., J. Biotechnol., 2012, 157(2), 315—319 |
| 26 | La R. G., Iaconelli M., Mancini P., Ferraro G. B., Veneri C., Bonadonna L., Lucentini L., Suffredini E., Sci. Total Environ., 2020, 736, 139652 |
| 27 | Sherchan S. P., Shahin S., Ward L. M., Tandukar S., Aw T. G., Schmitz B., Ahmed W., Kitajima M., Sci. Total Environ., 2020, 743, 140621 |
| 28 | Jafferali M. H., Khatami K., Atasoy M., Birgersson M., Williams C., Cetecioglu Z., Sci. Total Environ., 2021, 755, 142939 |
| 29 | Bar Y. M., Flamholz A., Phillips R., Milo R., eLife, 2020, 9, e57309 |
| 30 | Mousazadeh M., Ashoori R., Paital B., Kabdasli I., Frontistis Z., Hashemi M., Sandoval M. A., Sherchan S., Das K., Emamjomeh M. M., Pathogens, 2021, 10(8), 1008 |
| 31 | Qiu Y., Lee B. E., Ruecker N. J., Neumann N., Ashbolt N., Pang X., J. Virol. Methods, 2016, 237, 150—153 |
| 32 | Fang Y., Zhang H., Xie J., Lin M., Ying L., Pang P., Ji W., Radiology, 2020, 296(2), 115—117 |
| 33 | Ali N., Rampazzo R. C. P., Costa A. D. T., Krieger M. A., Biomed Res. Int., 2017, 9306564 |
| 34 | Chu A. W. H., Chan W. M., Ip J. D., Yip C. C. Y., Chan J. F. W., Yuen K. Y., To K. K. W., J. Clin. Virol., 2020, 129, 104519 |
| 35 | Ning B., Yu T., Zhang S., Huang Z., Tian D., Lin Z., Niu A., Golden N., Hensley K., Threeton B., Sci. Adv., 2021, 7(2), eabe3703 |
| 36 | Mondal S., Feirer N., Brockman M., Preston M. A., Teter S. J., Ma D., Goueli S. A., Moorji S., Saul B., Cali J. J., Sci. Total Environ., 2021, 795, 148834 |
| 37 | Tripathy S., Agarkar T., Talukdar A., Sengupta M., Kumar A., Ghosh S., Talanta, 2023, 252, 123809 |
| 38 | Ma Y. D., Chen Y. S., Lee G. B., Sens. Actuators B-Chem., 2019, 296, 126647 |
| 39 | Zong N., Gao Y., Chen Y., Luo X., Jiang X., Anal. Chem. 2022, 94(12), 5196—5203 |
| 40 | Gao S., Wang J., Li D., Li Y., Lou C., Zha E., Yue X., Tie Z. Z.. Int. J. Food Microbiol., 2020, 322, 108587 |
| 41 | Xi Y., Xu C. Z., Xie Z. Z., Zhu D. L., Dong J. M., Xiao G., Mol. Cell. Probes, 2019, 45, 8—13 |
| 42 | Chomczynski P., Sacchi N., Anal. Biochem., 1987, 162(1), 156—159 |
| 43 | Iovieno A., Miller D., Lonnen J., Kilvington S., Alfonso E. C., J. Clin. Microbiol., 2011, 49(1), 476—477 |
| 44 | Bai Y., Cui Y., Paoli G. C., Shi C., Wang D., Zhou M., Zhang L., Shi X., Colloid Surface B, 2016, 145, 257—266 |
| 45 | Sun N., Deng C., Liu Y., Zhao X., Tang Y., Liu R., Xia Q., Yan W., Ge G., J. Chromatogr. A, 2014, 1325, 31—39 |
| 46 | Chomczynski P., Sacchi N., Nat. Protoc., 2006, 1(2), 581—585 |
| 47 | Chu D. K., Pan Y., Cheng S. M., Hui K. P., Krishnan P., Liu Y., Ng D. Y., Wan C. K., Yang P., Wang Q., Clin. Chem., 2020, 66(4): 549-555 |
| 48 | Ambrosi C., Prezioso C., Checconi P., Scribano D., Sarshar M., Capannari M., Tomino C., Fini M., Garaci E., Palamara A. T., J. Virol. Methods, 2021, 287, 114008 |
| 49 | Barnes K. G., Lachenauer A. E., Nitido A., Siddiqui S., Gross R., Beitzel B., Siddle K. J., Freije C. A., Dighero K. B., Mehta S. B., Carter A., Uwanibe J., Ajogbasile F., Olumade T., Odia I., Sandi J. D., Momoh M., Metsky H. C., Boehm C. K., Lin A. E., Kemball M., Park D. J., Branco L., Boisen M., Sullivan B., Amare M. F., Tiamiyu A. B., Parker Z. F., Iroezindu M., Grant D. S., Modjarrad K., Myhrvold C., Garry R. F., Palacios G., Hensley L. E., Schaffner S. F., Happi C. T., Colubri A., Sabeti P. C., Nat. Commun., 2020, 11(1), 4131 |
| 50 | Arizti S. J., Freije C. A., Stanton A. C., Petros B. A., Boehm C. K., Siddiqui S., Shaw B. M., Adams G., Kosoko T. T. S. F., Kemball M. E., Nat. Commun., 2020, 11(1), 5921 |
| 51 | Odiwuor N., Xiong J., Ogolla F., Hong W., Li X., Khan F. M., Wang N., Yu J., Wei H., Anal. Chim. Acta, 2022, 1200, 339590 |
| 52 | Myhrvold C., Freije C. A., Gootenberg J. S., Abudayyeh O. O., Metsky H. C., Durbin A. F., Kellner M. J., Tan A. L., Paul L. M., Parham L. A., Garcia K. F., Barnes K. G., Chak B., Mondini A., Nogueira M. L., Isern S., Michael S. F., Lorenzana I., Yozwiak N. L., MacInnis B. L., Bosch I., Gehrke L., Zhang F., Sabeti P. C., Science, 2018, 360(6387), 444—448 |
| 53 | Mautner L., Baillie C. K., Herold H. M., Volkwein W., Guertler P., Eberle U., Ackermann N., Sing A., Pavlovic M., Goerlich O., Busch U., Wassill L., Huber I., Baiker A., Virol. J., 2020, 17(1), 160 |
| 54 | Cao G., Xiong Y., Qiu Y., Yang J., Wang Y., Nie F., Huo D., Hou C., Anal. Chim. Acta, 2023, 1280, 341865 |
| 55 | Krolov K., Frolova J., Tudoran O., Suhorutsenko J., Lehto T., Sibul H., Mager I., Laanpere M., Tulp I., Langel U., J. Mol. Diagn., 2014, 16(1), 127—135 |
| 56 | Notomi T., Okayama H., Masubuchi H., Yonekawa T., Watanabe K., Amino N., Hase T., Nucleic Acids Res., 2000, 28(12), e63 |
| 57 | Piepenburg O., Williams C. H., Stemple D. L., Armes N. A., PLoS Biol., 2006, 4(7), e204 |
| 58 | Tan E., Erwin B., Dames S., Ferguson T., Buechel M., Irvine B., Voelkerding K., Niemz A., Biochemistry, 2008, 47(38), 9987—9999 |
| 59 | Tomita N., Mori Y., Kanda H., Notomi T., Nat. Protoc., 2008, 3(5), 877—882 |
| 60 | Wei Z., Wang X., Feng H., Ji F., Bai D., Dong X., Huang W., Crit. Rev. Biotechnol., 2023, 43(3), 415—432 |
| 61 | Alhamid G., Tombuloglu H., Al-Suhaimi E., Sci. Rep., 2023, 13(1), 5066 |
| 62 | Yu Z., Lyu W., Yu M., Wang Q., Qu H., Ismagilov R. F., Han X., Lai D., Shen F., Biosens. Bioelectron., 2020, 155, 112107 |
| 63 | Lyu W., Zhang J., Yu Y., Xu L., Shen F., Lab Chip, 2021, 21(16), 3086—3093 |
| 64 | Yuan R., Wei J., Geng R., Li B., Xiong W., Fang X., Wang K., Sens. Actuators B-Chem., 2023, 380, 133363 |
| 65 | Crannell Z. A., Rohrman B., Richards K. R., Anal. Chem., 2014, 86(12), 5615—5619 |
| 66 | Gao X., Chen Y., Luo X., Du Z., Hao K., An M., Xia Z., Wu Y., ACS Omega, 2021, 6(28), 18008—18013 |
| 67 | Khater M., Escosura M. A., Altet L., Merkoci A., Anal. Chem., 2019, 91(7), 4790—4796 |
| 68 | Zhao C., Yang L., Zhang X., Tang Y., Wang Y., Shao X., Gao S., Liu X., Wang P., Anal. Chem., 2022, 94(49), 17151—17159 |
| 69 | Van N. J., Van N. L. K., Galas D. J., Proc. Natl. Acad. Sci. USA, 2003, 100(8), 4504—4509 |
| 70 | Carter J. G., Orueta I. L., Duprey J. H. A., Carter I. R., Southern C. D., Rana M., Whalley C. M., Bosworth A., Beggs A. D., Hicks M. R., Tucker J. H. R., Dafforn T. R., Proc. Natl. Acad. Sci. USA, 2021, 118(35), e2100347118 |
| 71 | Chen J., Zhu D., Huang T., Yang Z., Liu B., Sun M., Chen J. X., Dai Z., Zou X., Anal. Chem., 2021, 93(37), 12707—12713 |
| 72 | Compton J., Nature, 1991, 350(6313), 91—92 |
| 73 | Cordray M. S., Richards K. R. R., Am. J. Trop. Med. Hyg., 2012, 87(2), 223—230 |
| 74 | Walker G. T., Little M. C., Nadeau J. G., Shank D. D., Proc. Natl. Acad. Sci. USA, 1992, 89(1), 392—396 |
| 75 | Zhang C., Zheng T., Fan H., Zhang T., Han D., ACS Appl, Bio. Mater., 2021, 4(5), 3805—3810 |
| 76 | Daubendiek S. L., Ryan K., Kool E. T., J. Am. Chem. Soc., 1995, 117(29), 7818—7819 |
| 77 | Wang B., Potter S. J., Lin Y., Cunningham A. L., Dwyer D. E., Su Y., Ma X., Hou Y., Saksena N. K., J. Clin. Microbiol., 2005, 43(5), 2339—2344 |
| 78 | Vincent M., Xu Y., Kong H., Embo Rep., 2004, 5(8), 795—800 |
| 79 | Gaydos C. A., Schwebke J., Dombrowski J., Marrazzo J., Coleman J., Silver B., Barnes M., Crane L., Fine P., Expert Rev. Mol. Diagn., 2017, 17(3), 303—306 |
| 80 | Kowalski R. P., Karenchak L. M., Dhaliwal D. K., Mammen A., Eye Contact Lens., 2018, 44, S244—S248 |
| 81 | Barreda G. S., Miranda C. R., De L. S. A. N., Miranda O. A. J., Lobo C. M. J., Anal. Bioanal. Chem., 2018, 410(3), 679—693 |
| 82 | Li B., Ellington A. D., Chen X., Nucleic Acids. Res., 2011, 39(16), e110 |
| 83 | Dirks R. M., Pierce N. A., Proc. Natl. Acad. Sci. USA, 2004, 101(43), 15275—15278 |
| 84 | Li Y., Luo Z., Zhang C., Sun R., Zhou C., Sun C., TrAC-Trends Anal. Chem., 2021, 134, 116142 |
| 85 | Luo Z., Li Y., Zhang P., He L., Feng Y., Feng Y., Qian C., Tian Y., Duan Y., TrAC⁃Trends Anal. Chem., 2022, 151, 116582 |
| 86 | Yin P., Choi H. M., Calvert C. R., Pierce N. A., Nature, 2008, 451(7176), 318—322 |
| 87 | Zou M., Su F., Zhang R., Jiang X., Xiao H., Yan X., Yang C., Fan X., Wu G., Sens. Actuators B: Chem., 2021, 342(1), 129899 |
| 88 | Liang G. X., Ye S. Y., Yu H. M., Zhao K. R., Liu P. F., Liu Z. J., Wang L., Sens. Actuators B: Chem., 2022, 354, 131199 |
| 89 | Fu J., Wu J., Zhang R., Wu Q., Ju H., Sens. Actuators B: Chem., 2021, 345(15), 130436 |
| 90 | Lee H., Lee S., Park C., Yeom M., Lim J. W., Vu T. T. H., Kim E., Song D., Haam S., Small, 2023, 19(26), e2207117 |
| 91 | Fan Z., Yao B., Ding Y., Zhao J., Xie M., Zhang K., Biosens. Bioelectron., 2021, 178, 113015 |
| 92 | Zhang K., Fan Z., Ding Y., Zhu S., Xie M., Hao N., Environ. Sci.: Nano, 2022, 9(1), 162—172 |
| 93 | Jinek M., Chylinski K., Fonfara I., Hauer M., Doudna J. A., Charpentier E., Science, 2012, 337(6096), 816—821 |
| 94 | Huang M., Zhou X., Wang H., Xing D., Anal. Chem., 2018, 90(3), 2193—2200 |
| 95 | Santiago F. A., Hall L. N., Nemudraia A., Nemudryi A., Krishna P., Wiegand T., Wilkinson R. A., Snyder D. T., Hedges J. F., Cicha C., Cell Rep. Med., 2021, 2(6), 100319 |
| 96 | Chen J. S., Ma E., Harrington L. B., Da Costa. M., Tian X., Palefsky J. M., Doudna J. A., Science, 2018, 360(6387), 436—439 |
| 97 | Broughton J. P., Deng X., Yu G., Fasching C. L., Servellita V., Singh J., Miao X., Streithorst J. A., Granados A., Sotomayor G. A., Nat. Biotechnol., 2020, 38(7), 870—874 |
| 98 | Gootenberg J. S., Abudayyeh O. O., Lee J. W., Essletzbichler P., Dy A. J., Joung J., Verdine V., Donghia N., Daringer N. M., Freije C. A., Myhrvold C., Bhattacharyya R. P., Livny J., Regev A., Koonin E. V., Hung D. T., Sabeti P. C., Collins J. J., Zhang F., Science, 2017, 356(6336), 438—442 |
| 99 | Myhrvold C., Freije C. A., Gootenberg J. S., Abudayyeh O. O., Metsky H. C., Durbin A. F., Kellner M. J., Tan A. L., Paul L. M., Parham L. A., Science, 2018, 360(6387), 444—448 |
| 100 | Harrington L. B., Burstein D., Chen J. S., Paez E. D., Ma E., Witte I. P., Cofsky J. C., Kyrpides N. C., Banfield J. F., Doudna J. A., Science, 2018, 362(6416), 839—842 |
| 101 | Gootenberg J. S., Abudayyeh O. O., Kellner M. J., Joung J., Collins J. J., Zhang F., Science, 2018, 360(6387), 439—444 |
| 102 | Kellner M. J., Koob J. G., Gootenberg J. S., Abudayyeh O. O., Zhang F., Nat. Protoc., 2019, 14(10), 2986—3012 |
| 103 | Yang F., Wang W., Zhang M., Tao W., Wang Y., Shi J., Ding Y., Xie M., Zhang S., Fan Z., Zhang K., Environ. Sci.: Nano, 2022, 9(9), 3417—3426 |
| 104 | Kachwala M. J., Smith C. W., Nandu N., Yigit M. V., Anal. Chem., 2021, 93(4), 1934—1938 |
| 105 | Zhao R., Yu C., Lu B., Li B., RSC Adv., 2022, 12(17), 10374—10378 |
| 106 | Yang Y., Yi W., Gong F., Tan Z., Yang Y., Shan X., Xie C., Ji X., Zheng Z., He Z., Anal. Chem., 2023, 95(2), 1343—1349 |
| 107 | Ding X., Yin K., Li Z., Lalla R. V., Ballesteros E., Sfeir M. M., Liu C., Nat. Commun., 2020, 11(1), 4711 |
| 108 | Peng R., Lu Z., Liu M., Hu F., Sens. Actuators B: Chem., 2024, 399(15), 134838 |
| 109 | Joung J., Ladha A., Saito M., Kim N. G., Woolley A. E., Segel M., Barretto R. P., Ranu A., Macrae R. K., Faure G., N. Engl. J. Med., 2020, 383(15), 1492—1494 |
| 110 | Ghouneimy A., Ali Z., Aman R., Jiang W., Aouida M., Mahfouz M., ACS Synth. Biol., 2024, 13(3), 837—850 |
| 111 | Chen S., Wu C., Qian C., Pang Y., Guo K., Wang T., Bai L., Qian F., Ye Z., Liu Z., Qiao Z., Wang Y., Wang R., Anal. Chem., 2024, 96(7), 3145—3152 |
| 112 | Ooi K. H., Liu M. M., Tay J. W. D., Teo S. Y., Kaewsapsak P., Jin S., Lee C. K., Hou J., Maurer S. S., Lin W., Yan B.,Yan G., Gao Y. G., Tan M. H., Nat. Commun., 2021, 12(1), 1739 |
| 113 | Wang R., Qian C., Pang Y., Li M., Yang Y., Ma H., Zhao M., Qian F., Yu H., Liu Z., Biosens. Bioelectron., 2021, 172, 112766 |
| 114 | Pang B., Xu J., Liu Y., Peng H., Feng W., Cao Y., Wu J., Xiao H., Pabbaraju K., Tipples G., Anal. Chem., 2020, 92(24), 16204—16212 |
| 115 | Chen Y., Shi Y., Chen Y., Yang Z., Wu H., Zhou Z., Li J., Ping J., He L., Shen H., Biosens. Bioelectron., 2020, 169, 112642 |
| 116 | Gu W., Crawford E. D., O'Donovan B D., Wilson M. R., Chow E. D., Retallack H., DeRisi J. L., Genome. Biol., 2016, 17, 41 |
| 117 | Quan J., Langelier C., Kuchta A., Batson J., Teyssier N., Lyden A., Caldera S., McGeever A., Dimitrov B., King R., Wilheim J., Murphy M., Ares L. P., Travisano K. A., Sit R., Amato R., Mumbengegwi D. R., Smith J. L., Bennett A., Gosling R., Mourani P. M., Calfee C. S., Neff N. F., Chow E. D., Kim P. S., Greenhouse B., DeRisi J. L., Crawford E. D., Nucleic Acids. Res., 2019, 47(14), e83 |
| 118 | Zhou W., Hu L., Ying L., Zhao Z., Chu P. K., Yu X. F., Nat. Commun., 2018, 9(1), 5012 |
| 119 | Wang T., Liu Y., Sun H. H., Yin B. C., Ye B. C., Angew. Chem., Int. Ed. Engl., 2019, 58(16), 5382—5386 |
| 120 | Pardee K., Green A. A., Takahashi M. K., Braff D., Lambert G., Lee J. W., Ferrante T., Ma D., Donghia N., Fan M., Daringer N. M., Bosch I., Dudley D. M., O'connor D. H., Gehrke L., Collins J. J., Cell, 2016, 165(5), 1255—1266 |
| 121 | Oliveira B. B., Veigas B., Baptista P. V., Front. Sens., 2021, 2, 752600 |
| 122 | Morris U., Aydin S. B., Diagnostics, 2021, 11(2), 336 |
| 123 | Egerer R., Edel B., Loffler B., Henke A., Rodel J., J. Clin. Virol., 2021, 138, 104817 |
| 124 | De M. C., Koppelman M., Montes B., Ferre V., Soriano V., Cuypers H., Segondy M., Oosterlaken T., J. Virol. Methods, 2005, 127(1), 54—59 |
| 125 | Garrido C., Zahonero N., Corral A., Arredondo M., Soriano V., De M. C., J. Clin. Microbiol., 2009, 47(4), 1031—1036 |
| 126 | Saad A. A., Ahmed N. G., Osman O. S., Al B. A. A., Hamad A., Deborggraeve S., Buscher P., Schoone G. J., Schallig H. D., Laurent T., Haleem A., Osman O. F., Eltom A. M., Elbashir M. I., El S. S., PLoS Negl. Trop. Dis., 2010, 4(8), e776 |
| 127 | Carson C., Quinnell R. J., Holden J., Garcez L. M., Deborggraeve S., Courtenay O., J. Clin. Microbiol., 2010, 48(9), 3325—3330 |
| 128 | Capaul S. E., Gorgievski H. M., J. Clin. Virol., 2005, 32(3), 236—240 |
| 129 | Tong Y., Lemieux B., Kong H., BMC Biotechnol., 2011, 11(1), 1—7 |
| 130 | Noren T., Unemo M., Magnusson C., Eiserman M., Matussek A., APMIS, 2014, 122(2), 155—160 |
| 131 | Rakotosamimanana N., Lapierre S. G., Raharimanga V., Raherison M. S., Knoblauch A. M., Raherinandrasana A. H., Rakotoson A., Rakotonirina J., Rasolofo V., BMC Infect. Dis., 2019, 19(1), 542 |
| 132 | Lim H. S., Zheng Q., Miks K. M., Turner M., Yuk H. G., J. Food Prot., 2015, 78(6), 1203—1207 |
| 133 | Kurosaki Y., Magassouba N., Oloniniyi O. K., Cherif M. S., Sakabe S., Takada A., Hirayama K., Yasuda J., PLoS Negl. Trop. Dis., 2016, 10(2), e0004472 |
| 134 | Van D. P. B., Williams J. A., Fuller D., Taylor S. N., Hook E. W., J. Clin. Microbiol., 2017, 55(1), 155—164 |
| [1] | 赵雪琪, 赵越, 薛静, 白敏, 陈锋, 孙颖, 宋大千, 赵永席. 单细胞核酸编码扩增成像分析[J]. 高等学校化学学报, 2022, 43(12): 20220572. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||