

高等学校化学学报 ›› 2024, Vol. 45 ›› Issue (11): 20240299.doi: 10.7503/cjcu20240299
沈枫林1, 冯兆莹2, 方静1, 张磊1, 刘晓慧2( ), 周新文1(
), 周新文1( )
)
收稿日期:2024-06-21
出版日期:2024-11-10
发布日期:2024-09-11
通讯作者:
刘晓慧
E-mail:liuxiaohuijd@sjtu.edu.cn;zhouxinwen@fudan.edu.cn
作者简介:周新文, 男, 硕士, 高级工程师, 主要从事蛋白质组学方面的研究. E-mail: zhouxinwen@fudan.edu.cn基金资助:
SHEN Fenglin1, FENG Zhaoying2, FANG Jing1, ZHANG Lei1, LIU Xiaohui2( ), ZHOU Xinwen1(
), ZHOU Xinwen1( )
)
Received:2024-06-21
Online:2024-11-10
Published:2024-09-11
Contact:
LIU Xiaohui
E-mail:liuxiaohuijd@sjtu.edu.cn;zhouxinwen@fudan.edu.cn
Supported by:摘要:
细胞的异质性普遍存在, 单细胞及单细胞分辨的空间蛋白质组学的研究能够帮助人类更加深入地了解疾病机理. 然而, 受限于单细胞中蛋白含量低以及检测技术灵敏度等问题, 单细胞分辨的蛋白质组学的发展面临着一系列重大技术挑战. 近年来, 随着质谱仪器的灵敏度、 分辨率和扫描速度等方面的提升, 许多新的基于质谱的单细胞分辨率的空间蛋白组学新技术被开发出来, 并用于定量单个细胞中数千种的蛋白, 给疾病、 环境和医药等研究领域提供了高分辨的数据, 具有广阔的应用前景. 本文综合评述了这些基于质谱的单细胞分辨率的空间蛋白质组学的新技术, 并对其未来的发展趋势进行了展望.
中图分类号:
TrendMD:
沈枫林, 冯兆莹, 方静, 张磊, 刘晓慧, 周新文. 基于质谱的单细胞分辨的空间蛋白质组学新技术研究. 高等学校化学学报, 2024, 45(11): 20240299.
SHEN Fenglin, FENG Zhaoying, FANG Jing, ZHANG Lei, LIU Xiaohui, ZHOU Xinwen. New Technologies for Mass Spectrometry-based Single-cell Resolved Spatial Proteomics Research. Chem. J. Chinese Universities, 2024, 45(11): 20240299.
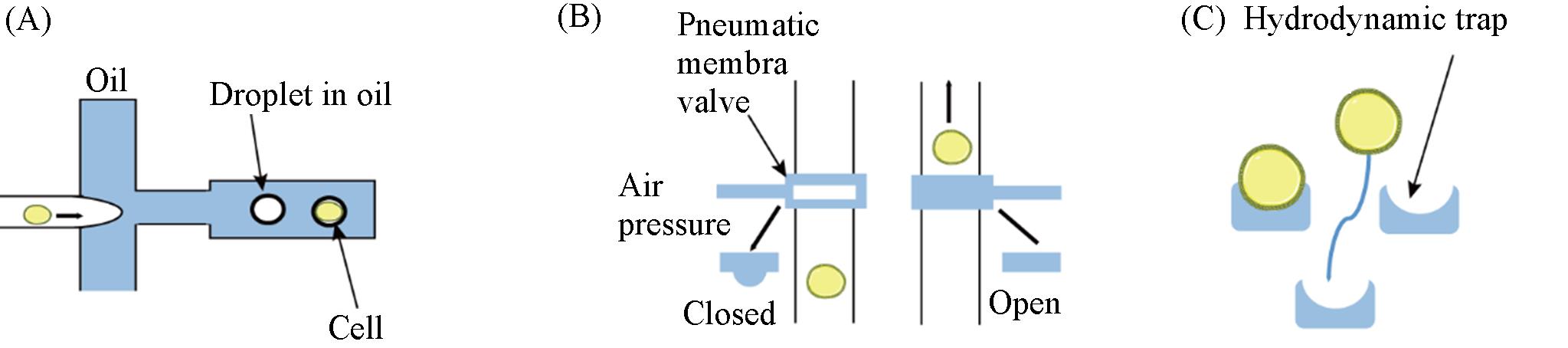
Fig.3 Schematic overview of different microfluidic methods for single⁃cell isolation(A) An aqueous stream of cells is broken up into individual droplets⁃in⁃oil containing random distribution of cells; (B) pneumatic membrane valves use air pressure to close a microfluidic channel by membrane deflection, this stops the flow and can trap a cell; (C) hydrodynamic traps are passive elements that only fit single cells and hold them at one position.
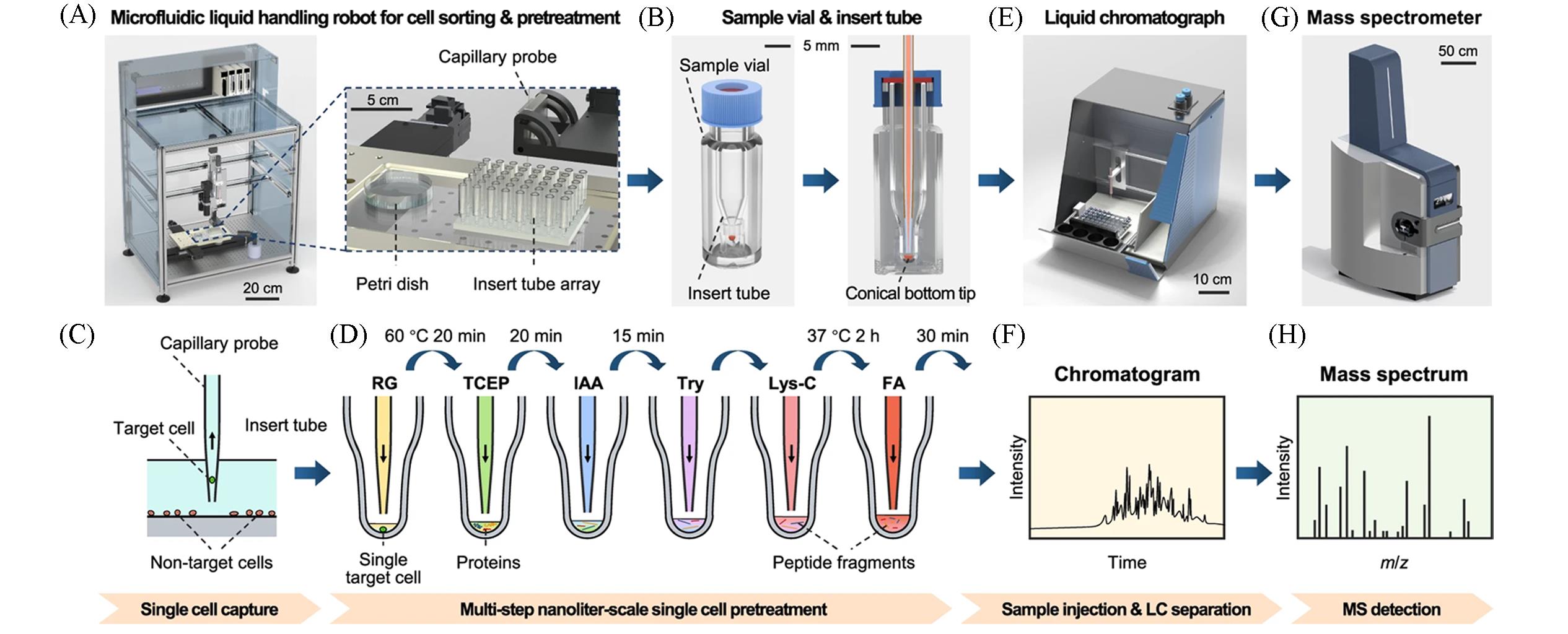
Fig.6 Schematic diagram of the PiSPA workflow[44](A) Microfluidic liquid handling robot with an insert tube array; (B) sorting of single cells and the multi⁃step pretreatment of the single cell samples; (C) sample preparation steps; (D) insert tubes coupled with sample vials were used as the nanoliter microreactors for sample pretreatment of single cell; (E) LC separation; (F) MS detection; (G) mass spectrometer; (H) mass spectrum.Copyright 2024, Nature Communications.
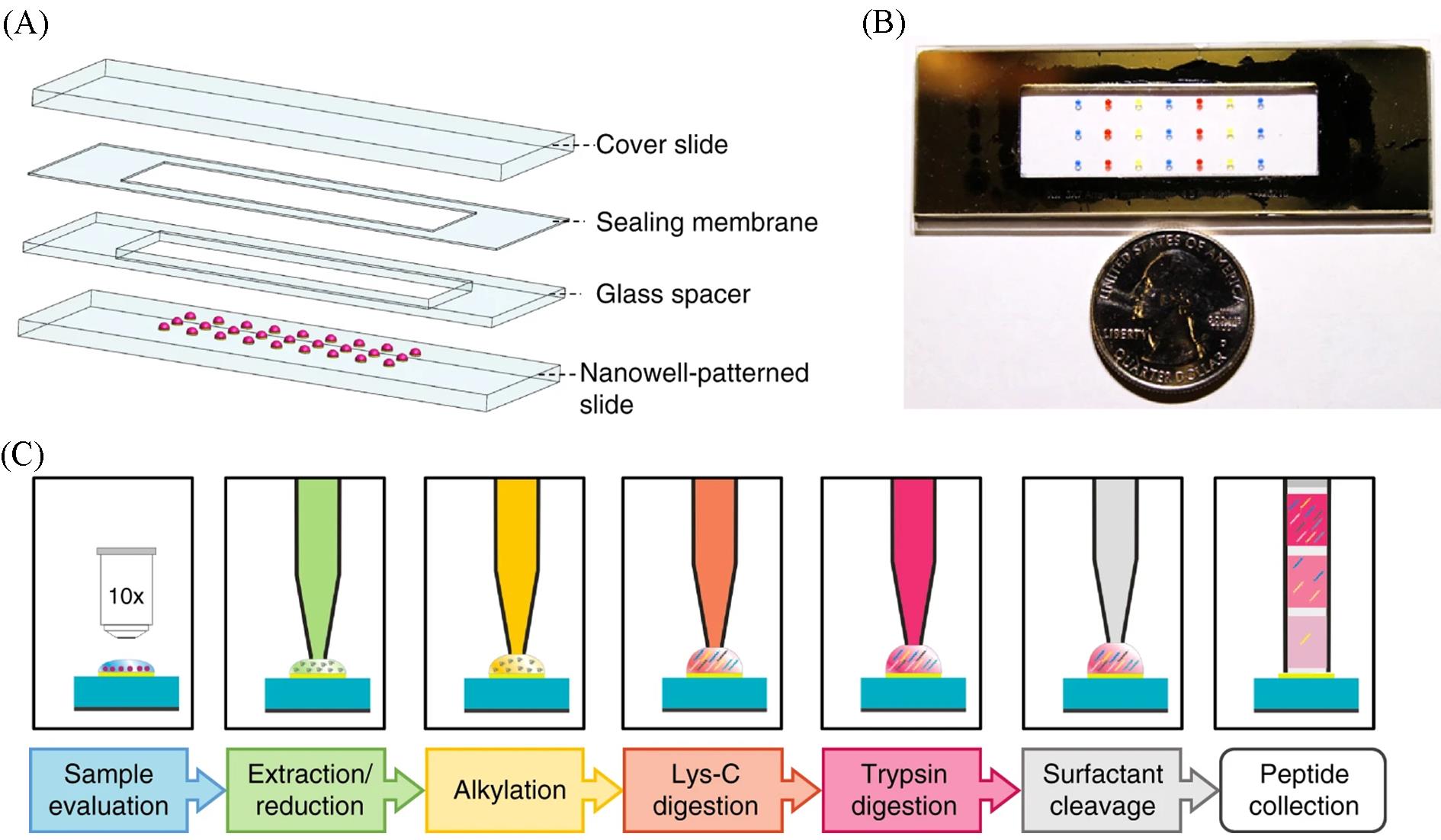
Fig.7 Proteomic sample preparation with nanoPOTs[45](A) Schematic drawing; (B) photograph showing the nanoPOTs chip with each nanowell filled with 200 nL of colored dye; (C) one⁃pot protocol for proteomic sample preparation and capillary⁃based sample collection.Copyright 2018, Nature Communications.
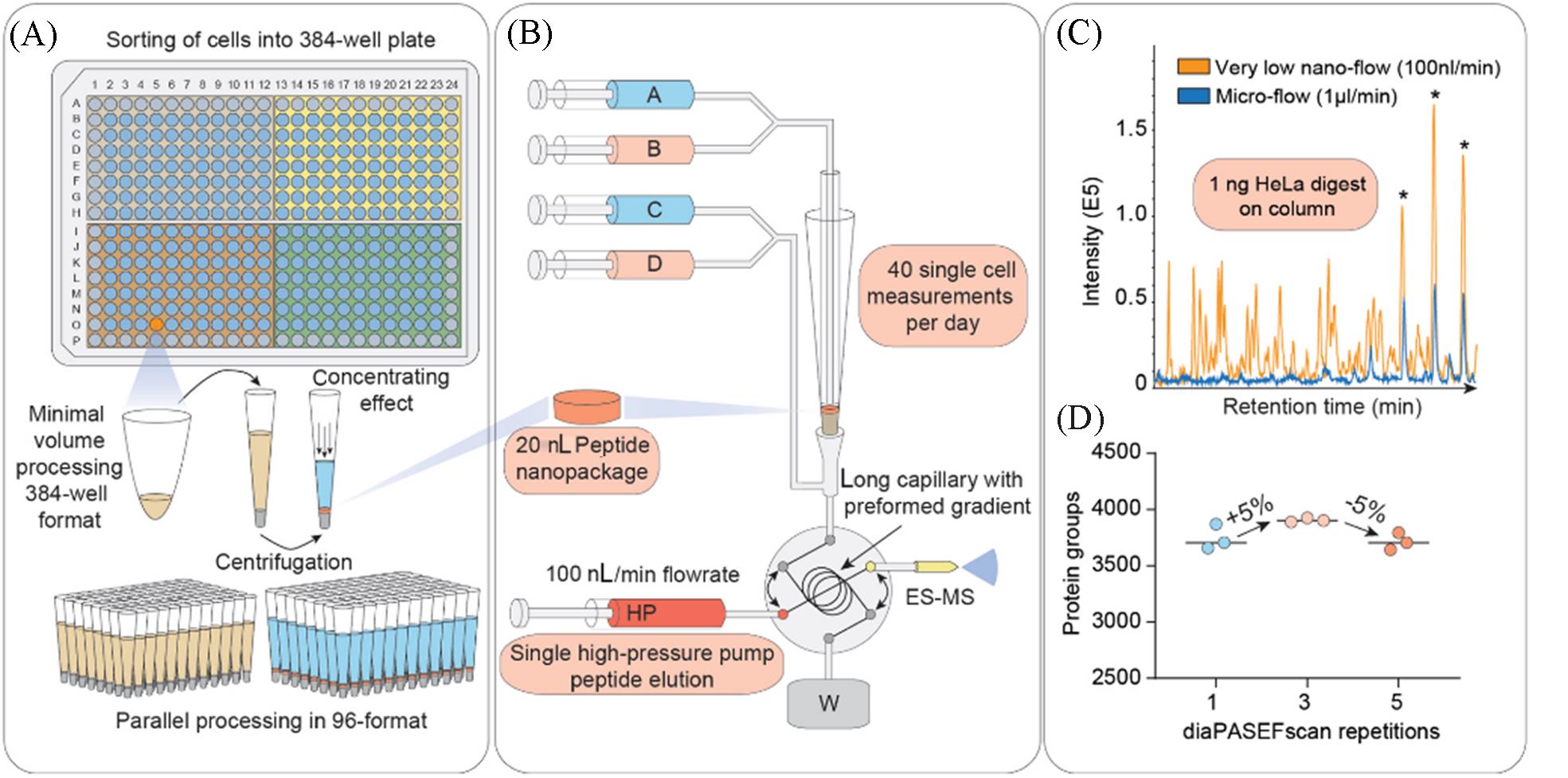
Fig.8 Schematic diagram of the T⁃SCP workflow[53](A) A Single cells are sorted in a 384⁃well format into 1 µL lysis buffer by FACS with outer wells serving as qualitative and quantitative controls; (B) these tips are automatically picked and peptide nanopackages are eluted in a sub⁃100⁃nL volume; (C) base⁃peak chromatogram of the standardized nanoflow(100 nL/min, orange) and microflow(1 µL/min, blue) gradients with 1 ng of HeLa digest on the StageTip; (D) nanoflow(100 nL/min) and short⁃gradient diaPASEF method combined.Copyright 2022, Molecular systems biology.
| 1 | Regev A., Teichmann S. A., Lander E. S., Amit I., Benoist C., Birney E., Bodenmiller B., Campbell P., Carninci P., Clatworthy M., Clevers H., Deplancke B., Dunham I., Eberwine J., Eils R., Enard W., Farmer A., Fugger L., Gottgens B., Hacohen N., Haniffa M., Hemberg M., Kim S., Klenerman P., Kriegstein A., Lein E., Linnarsson S., Lundberg E., Lundeberg J., Majumder P., Marioni J. C., Merad M., Mhlanga M., Nawijn M., Netea M., Nolan G., Pe'er D., Phillipakis A., Ponting C. P., Quake S., Reik W., Rozenblatt⁃Rosen O., Sanes J., Satija R., Schumacher T. N., Shalek A., Shapiro E., Sharma P., Shin J. W., Stegle O., Stratton M., Stubbington M. J. T., Theis F. J., Uhlen M., van Oudenaarden A., Wagner A., Watt F., Weissman J., Wold B., Xavier R., Yosef N., Human Cell Atlas Meeting, P. Elife, 2017, 6 |
| 2 | Kaern M., Elston T. C., Blake W. J., Collins J. J., Nat. Rev. Genet., 2005, 6(6), 451—464 |
| 3 | Altschuler S. J., Wu L. F., Cell, 2010, 141(4), 559—563 |
| 4 | Nat. Methods, 2021, 18(1), 1 |
| 5 | Tang F., Barbacioru C., Wang Y., Nordman E., Lee, C., Xu N., Wang X., Bodeau J., Tuch B. B., Siddiqui A., Lao K., Surani M. A., Nat. Methods, 2009, 6(5), 377—382 |
| 6 | Mamlouk S., Childs L. H., Aust D., Heim D., Melching F., Oliveira C., Wolf T., Durek P., Schumacher D., Blaker H., von Winterfeld M., Gastl B., Mohr K., Menne A., Zeugner S., Redmer T., Lenze D., Tierling S., Mobs M., Weichert W., Folprecht G., Blanc E., Beule D., Schafer R., Morkel M., Klauschen F., Leser U., Sers C., Nat. Commun., 2017, 8, 14093 |
| 7 | Aebersold R., Mann M., Nature, 2016, 537(7620), 347—355 |
| 8 | Cui L., Lu H., Lee Y. H., Mass Spectrom. Rev., 2018, 37(6), 772—792 |
| 9 | Lanekoff I., Sharma V. V., Marques C., Curr. Opin. Biotechnol., 2022, 75, 102693 |
| 10 | Davis S., Scott C., Oetjen J., Charles P. D., Kessler B. M., Ansorge O., Fischer R., Nat. Commun., 2023, 14(1), 7710 |
| 11 | Vistain L. F., Tay S., Trends Biochem. Sci., 2021, 46(8), 661—672 |
| 12 | Yang S., Xiong Y., Du Y., Wang Y. J., Zhang L., Shen F., Liu Y. J., Liu X., Yang P., Anal. Chem., 2022, 94(2), 768—776 |
| 13 | Arias⁃Hidalgo C., Juanes⁃Velasco P., Landeira⁃Vinuela A., Garcia⁃Vaquero M. L., Montalvillo E., Gongora R., Hernandez A. P., Fuentes M., Int. J. Mol. Sci., 2022, 23(12), 6707—6725. |
| 14 | Melby J. A., Brown K. A., Gregorich Z. R., Roberts D. S., Chapman E. A., Ehlers L. E., Gao Z., Larson E. J., Jin Y., Lopez J. R., Hartung J., Zhu Y., McIlwain S. J., Wang D., Guo W., Diffee G. M., Ge Y., Proc. Natl. Acad. Sci. USA, 2023, 120(19), e2222081120 |
| 15 | Johnson K. R., Gao Y., Gregus M., Ivanov A. R., Anal. Chem., 2022, 94(41), 14358—14367 |
| 16 | Brown M., Wittwer C., Clin. Chem., 2000, 46(8 Pt 2), 1221—1229 |
| 17 | Valet G., J. Biol. Regul. Homeost. Agents., 2003, 17(3), 213—222 |
| 18 | Davey H. M., Kell D. B., Microbiol. Rev., 1996, 60(4), 641—696 |
| 19 | Lacombe F., Belloc F., Hematol. Cell Ther., 1996, 38(6), 495—504 |
| 20 | McCoy J. P. Jr., Carey J. L., Immunol. Ser., 1990, 53, 171—187 |
| 21 | Fröhlich J., König H., Fems. Microbiol. Rev., 2000, 24(5), 567—572 |
| 22 | Wright G., Tucker M. J., Morton P. C., Sweitzer⁃Yoder C. L., Smith S. E., Curr. Opin. Obstet. Gyn., 1998, 10(3), 221—226 |
| 23 | Emmert⁃Buck M. R., Bonner R. F., Smith P. D., Chuaqui R. F., Zhuang Z., Goldstein S. R., Weiss R. A., Liotta L. A., Science, 1996, 274(5289), 998—1001 |
| 24 | Liu A., JBT., 2010, 21(3), 120—125 |
| 25 | Mund A., Coscia F., Kriston A., Hollandi R., Kovacs F., Brunner A. D., Migh E., Schweizer L., Santos A., Bzorek M., Naimy S., Rahbek⁃Gjerdrum L. M., Dyring⁃Andersen B., Bulkescher J., Lukas C., Eckert M. A., Lengyel E., Gnann C., Lundberg E., Horvath P., Mann M., Nat. Biotechnol., 2022, 40(8), 1231—1240 |
| 26 | Hu P., Zhang W., Xin H., Deng G., Front. Cell Dev. Biol., 2016, 4, 116 |
| 27 | Bandura D. R., Baranov V. I., Ornatsky O. I., Antonov A., Kinach R., Lou X., Pavlov S., Vorobiev S., Dick J. E., Tanner S. D., Anal. Chem., 2009, 81(16), 6813—6822 |
| 28 | Yoshida Y., Yoshio S., Yamazoe T., Mori T., Tsustui Y., Kawai H., Yoshikawa S., Fukuhara T., Okamoto T., Ono Y., Takahashi Y., Hashida R., Kawaguchi T., Taketomi A., Kanto T., Cells, 2021, 10(6), 1495 |
| 29 | Spitzer M. H., Gherardini P. F., Fragiadakis G. K., Bhattacharya N., Yuan R. T., Hotson A. N., Finck R., Carmi Y., Zunder E. R., Fantl W. J., Bendall S. C., Engleman E. G., Nolan G. P., Science, 2015, 349(6244), 1259425 |
| 30 | Bodenmiller B., Zunder E. R., Finck R., Chen T. J., Savig E. S., Bruggner R. V., Simonds E. F., Bendall S. C., Sachs K., Krutzik P. O., Nolan G. P., Nat. Biotechnol., 2012, 30(9), 858—867 |
| 31 | Krishnaswamy S., Spitze, M. H., Mingueneau M., Bendall S. C., Litvin O., Stone E., Pe'er D., Nolan G. P., Science, 2014, 346(6213), 1250689 |
| 32 | Giesen C., Wang H. A., Schapiro D., Zivanovic N., Jacobs A., Hattendorf B., Schuffler P. J., Grolimund D., Buhmann J. M., Brandt S., Varga Z., Wild P. J., Gunther D., Bodenmiller B., Nat. Methods, 2014, 11(4), 417—422 |
| 33 | Lohani V., Anikumar R. A., Kundu S., Akhter M. Q., Bag S., ACS Omega, 2023, 8(20), 17499—17510 |
| 34 | Norris J. L., Caprioli R. M., Chem. Rev., 2013, 113(4), 2309—2342 |
| 35 | Flatley B., Malone P., Cramer R., Biochimica. Et. biophysica. Acta., 2014, 1844(5), 940—949 |
| 36 | Schnackenberg L. K., Thorn D. A., Barnette D., Jones E. E., Metab. Brain. Dis., 2022, 37(1), 105—121 |
| 37 | Zhu X., Xu T., Peng C., Wu S., Front. Chem., 2021, 9, 782432 |
| 38 | Moginger U., Marcussen N., Jensen O. N., Oncotarget, 2020, 11(44), 3998—4015 |
| 39 | Hoffmann F., Umbreit C., Kruger T., Pelzel D., Ernst G., Kniemeyer O., Guntinas⁃Lichius O., Berndt A., von Eggeling F., Proteomics Clin. Appl., 2019, 13(1), e1700173 |
| 40 | Malaker S. A., Quanico J., Raffo⁃Romero A., Kobeissy F., Aboulouard S., Tierny D., Bertozzi C. R., Fournier I., Salzet M., Cell Chem. Biol., 2022, 29(1), 30—42 e34 |
| 41 | Kompauer M., Heiles S., Spengler B., Nat. Methods, 2017, 14(1), 90—96 |
| 42 | Mund A., Brunner A. D., Mann M., Mol. Cell, 2022, 82(12), 2335—2349 |
| 43 | Li Z. Y., Huang M., Wang X. K., Zhu Y., Li J. S., Wong C. C. L., Fang Q., Anal. Chem., 2018, 90(8), 5430—5438 |
| 44 | Wang Y., Guan Z. Y., Shi S. W., Jiang Y. R., Zhang J., Yang Y., Wu Q., Wu J., Chen J. B., Ying W. X., Xu Q. Q., Fan Q. X., Wang H. F., Zhou L., Wang L., Fang J., Pan J. Z., Fang Q., Nat. Commun., 2024, 15(1), 1279 |
| 45 | Zhu Y., Piehowski P. D., Zhao R., Chen J., Shen Y., Moore R. J., Shukla A. K., Petyuk V. A., Campbell⁃Thompson M., Mathews C. E., Smith R. D., Qian W. J., Kelly R. T., Nat. Commun., 2018, 9(1), 882 |
| 46 | Zhu Y., Clair G., Chrisler W. B., Shen Y., Zhao R., Shukla A. K., Moore R. J., Misra R. S., Pryhuber G. S., Smith R. D., Ansong C., Kelly R. T., Angew. Chem. Int. Ed. Engl., 2018, 57(38), 12370—12374 |
| 47 | Truong T., Webber K. G. I., Madisyn Johnston S., Boekweg H., Lindgren C. M., Liang Y., Nydegger A., Xie X., Tsang T. M., Jayatunge D., Andersen J. L., Payne S. H., Kelly R. T., Angew. Chem. Int. Ed. Engl., 2023, 62(34), e202303415 |
| 48 | Zhu Y., Dou M., Piehowski P. D., Liang Y., Wang F., Chu R. K., Chrisler W. B., Smith J. N., Schwarz K. C., Shen Y., Shukla A. K., Moore R. J., Smith R. D., Qian W. J., Kelly R. T., Mol. Cell Proteomics, 2018, 17(9), 1864—1874 |
| 49 | Xu K., Liang Y., Piehowski P. D., Dou M., Schwarz K. C., Zhao R., Sontag R. L., Moore R. J., Zhu Y., Kelly R. T., Anal. Bioanal. Chem., 2019, 411(19), 4587—4596 |
| 50 | Liang Y., Zhu Y., Dou M., Xu K., Chu R. K., Chrisler W. B., Zhao R., Hixson K. K., Kelly R. T., Anal. Chem., 2018, 90(18), 11106—11114 |
| 51 | Gebreyesus S. T., Siyal A. A., Kitata R. B., Chen E. S., Enkhbayar B., Angata, T., Lin K. I., Chen Y. J., Tu H. L., Nat. Commun., 2022, 13(1), 37 |
| 52 | Yang Z., Jin K., Chen Y., Liu Q., Chen H., Hu S., Wang Y., Pan Z., Feng F., Shi M., Xie H., Ma H., Zhou H., JACS Au, 2024, 4(5), 1811—1823 |
| 53 | Brunner A. D., Thielert M., Vasilopoulou C., Ammar C., Coscia F., Mund A., Hoerning O. B., Bache N., Apalategui A., Lubeck M., Richter S., Fischer D. S., Raether O., Park M. A., Meier F., Theis F. J., Mann M., Mol. Syst. Biol., 2022, 18(3), e10798 |
| 54 | Specht H., Harmange G., Perlman D. H., Emmott E., Niziolek Z., Budnik B., Slavov N., bioRxiv., 2018, doi: org/10.1101/399774. |
| 55 | Leduc A., Huffman R. G., Cantlon J., Khan S., Slavov N., Genome Biol., 2022, 23(1), 261—290 |
| 56 | Ye Z., Sabatier P., Martin⁃Gonzalez J., Eguchi A., Lechner M., Ostergaard O., Xie J., Guo Y., Schultz L., Truffer R., Bekker⁃Jensen D. B., Bache N., Olsen J. V., Nat. Commun., 2024, 15(1), 2474 |
| 57 | Shao X., Wang X., Guan S., Lin H., Yan G., Gao M., Deng C., Zhang X., Anal. Chem., 2018, 90(23), 14003—14010 |
| 58 | Masuda T., Inamori Y., Furukawa A., Yamahiro M., Momosaki K., Chang C. H., Kobayashi D., Ohguchi H., Kawano Y., Ito S., Araki N., Ong S. E., Ohtsuki S., Anal. Chem., 2022, 94(29), 10329—10336 |
| 59 | Tsai C. F., Zhang P., Scholten D., Martin K., Wang Y. T., Zhao R., Chrisler W. B., Patel D. B., Dou M., Jia Y., Reduzzi C., Liu X., Moore R. J., Burnum⁃Johnson K. E., Lin M. H., Hsu C. C., Jacobs J. M., Kagan J., Srivastava S., Rodland K. D., Steven Wiley H., Qian W. J., Smith R. D., Zhu Y., Cristofanilli M., Liu T., Liu H., Shi T., Commun. Biol., 2021, 4(1), 265 |
| 60 | Yang S., Han Y., Li Y., Zhang L., Yan G., Yuan J., Luo Q., Shen H., Liu X., Anal. Chem., 2023, 95(28), 10703—10712 |
| 61 | Bhatia H. S., Brunner A. D., Ozturk F., Kapoor S., Rong Z., Mai H., Thielert M., Ali M., Al⁃Maskari R., Paetzold J. C., Kofler F., Todorov M. I., Molbay M., Kolabas Z. I., Negwer M., Hoeher L., Steinke H., Dima A., Gupta B., Kaltenecker D., Caliskan O. S., Brandt D., Krahmer N., Muller S., Lichtenthaler S. F., Hellal F., Bechmann I., Menze B., Theis F., Mann M., Erturk A., Cell, 2022, 185(26), 5040—5058 |
| 62 | Meding S., Martin K., Gustafsson O. J., Eddes J. S., Hack S., Oehler M. K., Hoffmann P., J. Proteome Res., 2013, 12(1), 308—315 |
| 63 | Rauser S., Marquardt C., Balluff B., Deininger S. O., Albers C., Belau E., Hartmer R., Suckau D., Specht K., Ebert M. P., Schmitt M., Aubele M., Hofler H., Walch A., J. Proteome Res., 2010, 9(4), 1854—1863 |
| 64 | Piehowski P. D., Zhu Y., Bramer L. M., Stratton K. G., Zhao R., Orton D. J., Moore R. J., Yuan J., Mitchell H. D., Gao Y., Webb⁃Robertson B. M., Dey S. K., Kelly R. T., Burnum⁃Johnson K. E., Nat. Commun., 2020, 11(1), 8 |
| 65 | Li L., Sun C., Sun Y., Dong Z., Wu R., Sun X., Zhang H., Jiang W., Zhou Y., Cen X., Cai S., Xia H., Zhu Y., Guo T., Piatkevich K. D., Nat. Commun., 2022, 13(1), 7242 |
| 66 | Thielert M., Itang E. C., Ammar C., Rosenberger F. A., Bludau I., Schweizer L., Nordmann T. M., Skowronek P., Wahle M., Zeng W. F., Zhou X. X., Brunner A. D., Richter S., Levesque M. P., Theis F. J., Steger M., Mann M., Mol. Syst. Biol., 2023, 19(9), e11503 |
| 67 | Rosenberger F. A., Thielert M., Strauss M. T., Schweizer L., Ammar, C., Madler S. C., Metousis A., Skowronek P., Wahle M., Madden K., Gote⁃Schniering J., Semenova A., Schiller H. B., Rodriguez E., Nordmann T. M., Mund A., Mann M., Nat. Methods, 2023, 20(10), 1530—1536 |
| 68 | Budnik B., Levy E., Harmange G., Slavov N., Genome. Biol., 2018, 19(1), 161 |
| 69 | Petelski A. A., Emmott E., Leduc A., Huffman R. G., Specht H., Perlman D. H., Slavov N., Nat. Protoc., 2021, 16(12), 5398—5425 |
| 70 | Amstalden van Hove E. R., Smith D. F., Heeren R. M., J. Chromatogr. A, 2010, 1217(25), 3946—3954 |
| 71 | Vaysse P. M., Heeren R. M. A., Porta T., Balluff B., Analyst, 2017, 142(15), 2690—2712 |
| [1] | 黄玉滢, 于成鲲, 刘斯奇, 任艳. 利用无标记单细胞蛋白质组学方法构建小鼠外周血单个核细胞的细胞图谱[J]. 高等学校化学学报, 2024, 45(11): 20240355. |
| [2] | 霍志远, 周金萍, 马秀敏, 周严, 黄琳. 基于质谱的单细胞多组学分析技术研究进展[J]. 高等学校化学学报, 2024, 45(11): 20240389. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||