

高等学校化学学报 ›› 2024, Vol. 45 ›› Issue (11): 20240294.doi: 10.7503/cjcu20240294
范智瑞1, 方群1,2, 杨奕1,2
收稿日期:2024-06-19
出版日期:2024-11-10
发布日期:2024-08-19
通讯作者:
杨奕
作者简介:方 群, 男, 博士, 教授, 主要从事微流控、 色谱、 质谱和单细胞多组学方面的研究. E-mail: fangqun@zju. edu. cn
基金资助:FAN Zhirui1, FANG Qun1,2, YANG Yi1,2
Received:2024-06-19
Online:2024-11-10
Published:2024-08-19
Contact:
YANG Yi
Supported by:摘要:
单细胞蛋白质组学分析能揭示细胞个体之间蛋白质的精细差异, 在诸多重要领域具有重要的应用价值, 已成为目前的研究热点; 其难点在于单细胞内的蛋白质极其微量, 需要解决样品处理过程中的损失问题、 色谱质谱检测的灵敏度问题和低信号强度质谱数据的解析利用问题. 本文从单细胞分选、 样品处理、 色谱质谱采集和数据分析等方面, 综合评述了目前基于质谱的单细胞蛋白质组学分析方法的研究进展及其在生物医学领域的应用, 并展望了其未来的发展前景.
中图分类号:
TrendMD:
范智瑞, 方群, 杨奕. 基于质谱的单细胞蛋白质组学分析. 高等学校化学学报, 2024, 45(11): 20240294.
FAN Zhirui, FANG Qun, YANG Yi. Single Cell Proteomic Analysis by Mass Spectrometry. Chem. J. Chinese Universities, 2024, 45(11): 20240294.
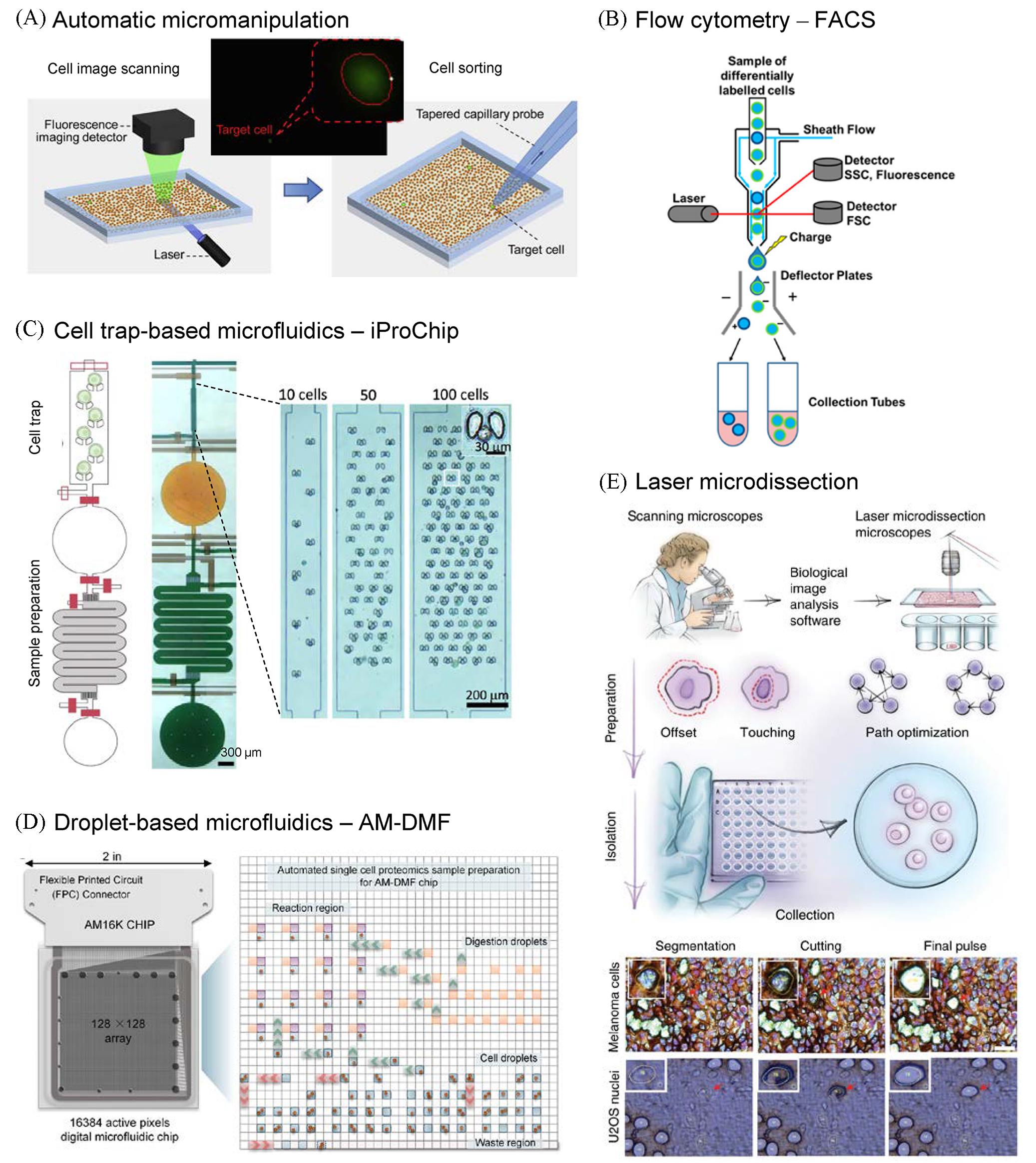
Fig.1 Single cell sorting methods(A) Automatic micromanipulation by machine vision[8]; (B) fluorescence-activated cell sorting(FACS)[12]; (C) integrated proteomics chip(iProChip)[16]; (D) active matrix digital microfluidic chip(AMD-MF)[18]; (E) deep visual proteomics by artificial intelligence-guided cell classification and laser microdissection[21]. (A) Copyright 2021, Elsevier; (B) Copyright 2015, MDPI; (C) Copyright 2021, Springer Nature; (D) Copyright 2024, American Chemical Society; (E) Copyright 2022, Springer Nature.
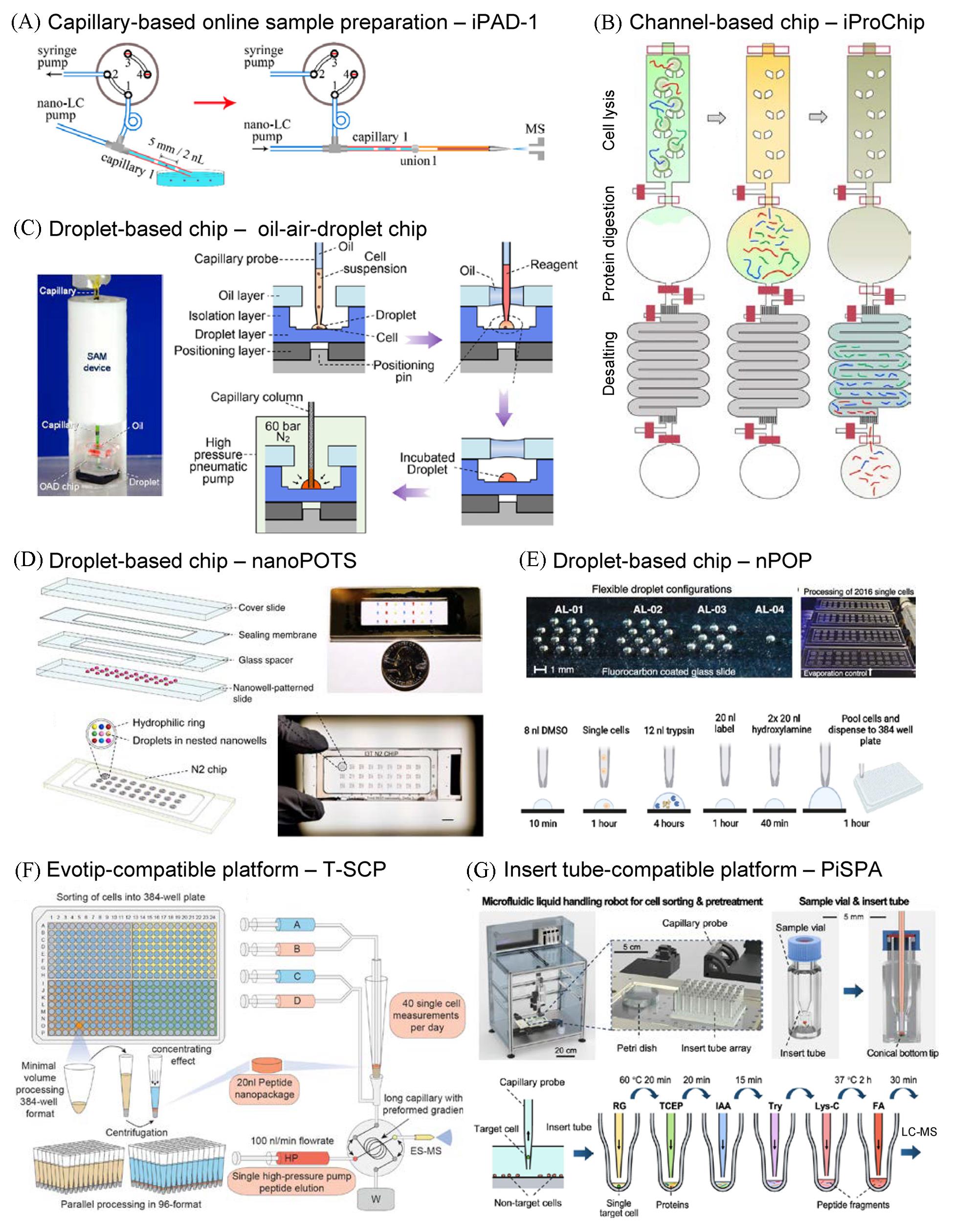
Fig.2 Single cell proteome sample preparation methods(A) Integrated proteome analysis device(iPAD-1)[25]; (B) integrated proteomics chip (iProChip)[16]; (C) oil-air-droplet chip[30]; (D) nanodroplet processing in one pot for trace samples(nanoPOTS)[31] and nested nanoPOTS(N2) chips[33]; (E) nano-proteomic sample preparation(nPOP)[35]; (F) true single-cell proteome(T-SCP) platform[37]; (G) pick-up single-cell proteomic analysis(PiSPA) platform[9]. (A) Copyright 2018, American Chemical Society; (B) Copyright 2021, Springer Nature; (C) Copyright 2018, American Chemical Society; (D) Copyright 2018, Springer Nature; Copyright 2021, the authors; (E) Copyright 2022, Springer Nature; (F) Copyright 2022, EMBO; (G) Copyright 2024, Springer Nature.
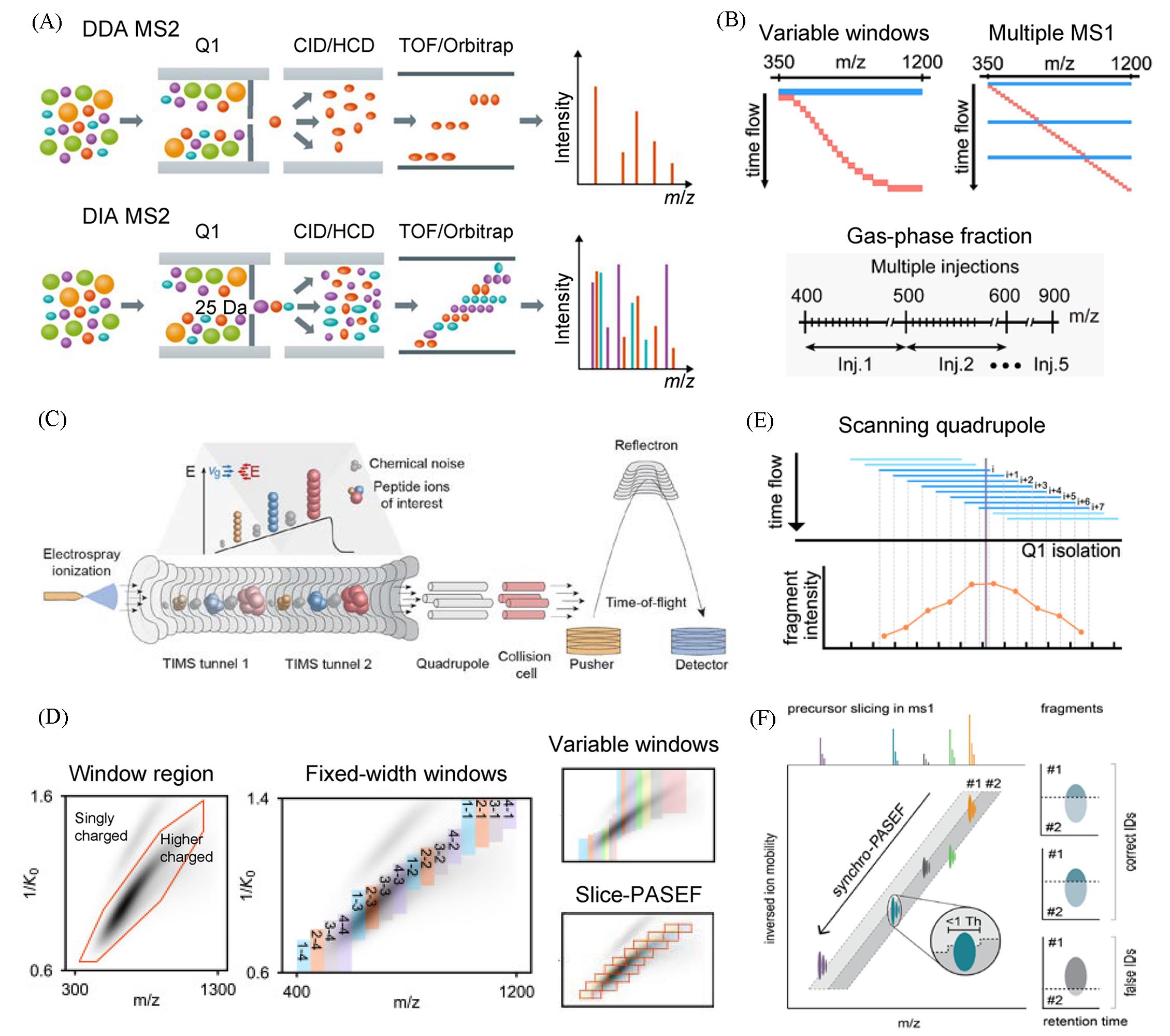
Fig.3 Mass spectrometry data acquisition methods(A) Data-dependent acquisition(DDA) and data-independent acquisition(DIA)[52]; (B) variant DIA schemes[55]; (C) DIA with parallel accumulation-serial fragmentation(diaPASEF)[37]; (D) various window arrangements for diaPASEF[55]; (E) scanning quadrupole DIA[55]; (F) Synchro-PASEF, where quadrupole scanning is synchronized with ion release in the mobility dimension[70]. (A) Copyright 2018, EMBO; (B, D, E) Copyright 2024, Elsevier; (C) Copyright 2022, EMBO; (F) Copyright 2023, Elsevier.
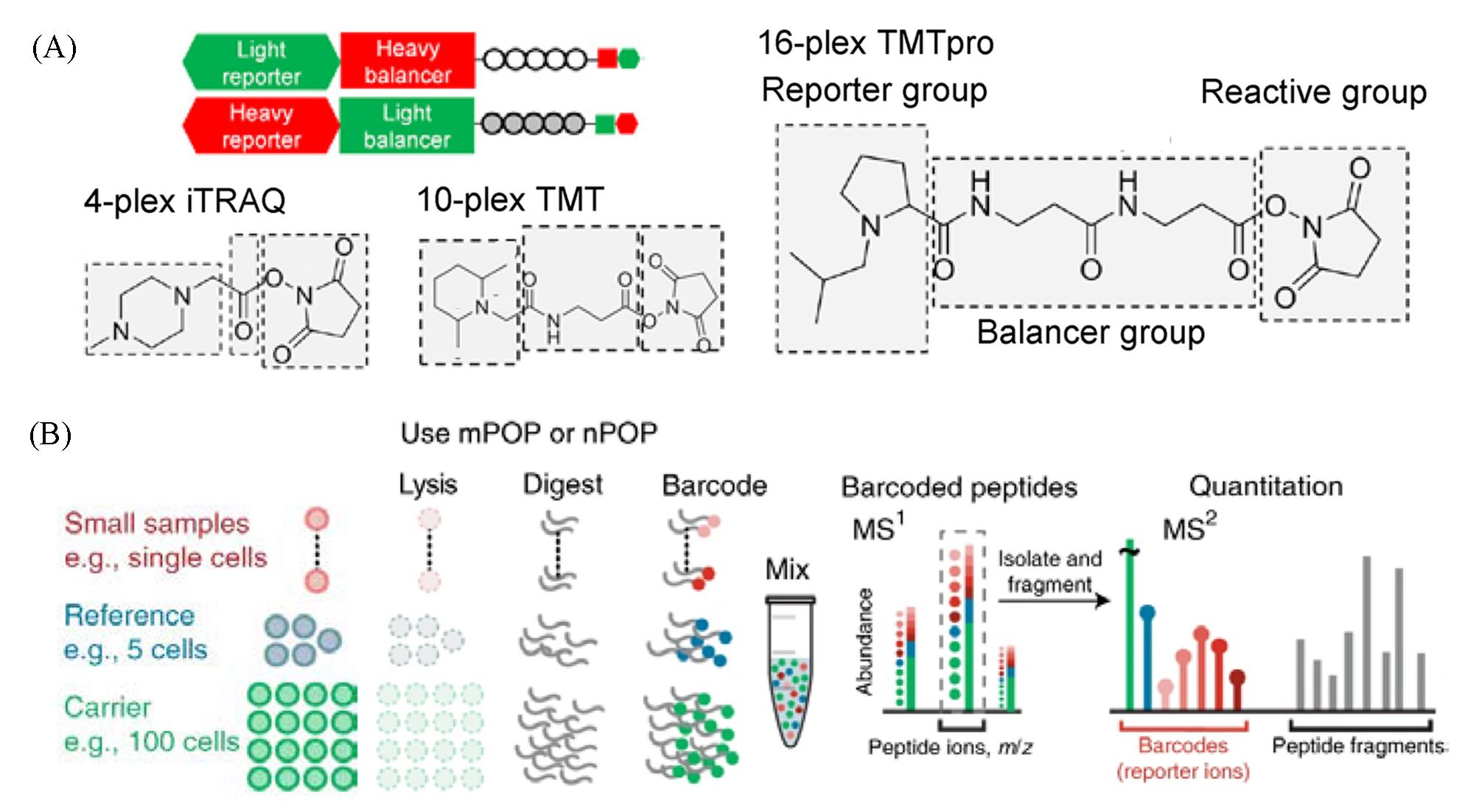
Fig.4 Isobaric labeling for reporter ion⁃based quantification(A) Representative labeling regents[72]; (B) multiplexed single-cell proteomics using SCoPE2[77]. (A) Copyright 2021, Wiley; (B) Copyright 2021, Springer Nature.
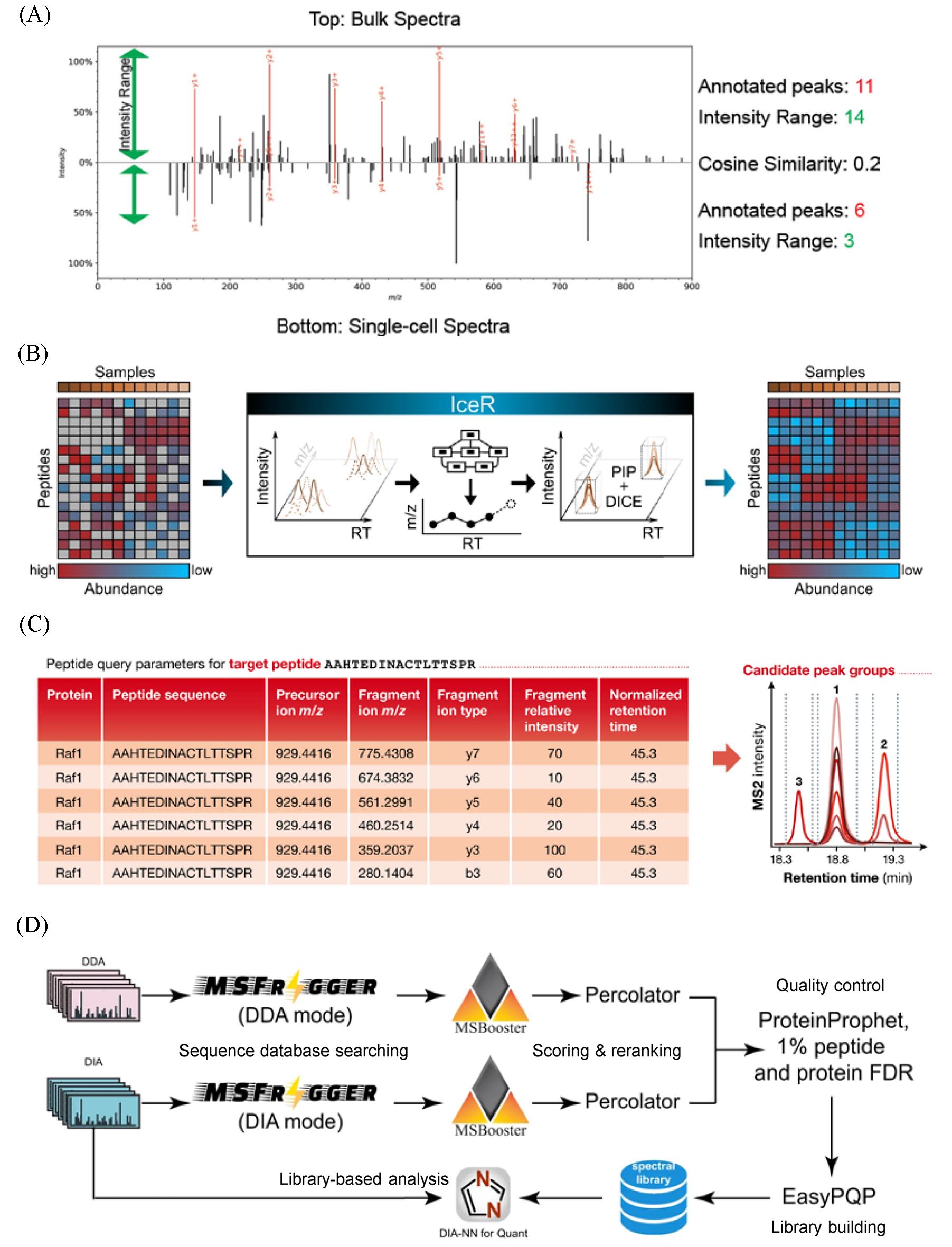
Fig.5 Mass spectrometry data analysis methods for proteomics(A) Spectral features of single-cell proteome samples[88]; (B) match-between-runs by IceR for reducing missing values[93]; (C) library-based DIA analysis by targeted data extraction[52]; (D) integrated DIA analysis workflow by FragPipe and DIA-NN[112]. (A) Copyright 2022, American Chemical Society; (B) Copyright 2021, Springer Nature; (C) Copyright 2018, EMBO; (D) Copyright 2023, Springer Nature.
| 1 | Wu Q., Sui X. T., Tian R. J., Chin. J. Chromatogr., 2021, 39(2), 112—117 |
| 吴琼, 隋欣桐, 田瑞军. 色谱, 2021, 39(2), 112—117 | |
| 2 | Regev A., Teichmann S. A., Lander E. S., Amit I., Benoist C., Birney E., Bodenmiller B., Campbell P., Carninci P., Clatworthy M., Clevers H., Deplancke B., Dunham I., Eberwine J., Eils R., Enard W., Farmer A., Fugger L., Göttgens B., Hacohen N., Haniffa M., Hemberg M., Kim S., Klenerman P., Kriegstein A., Lein E., Linnarsson S., Lundberg E., Lundeberg J., Majumder P., Marioni J. C., Merad M., Mhlanga M., Nawijn M., Netea M., Nolan G., Pe'er D., Phillipakis A., Ponting C. P., Quake S., Reik W., Rozenblatt⁃Rosen O., Sanes J., Satija R., Schumacher T. N., Shalek A., Shapiro E., Sharma P., Shin J. W., Stegle O., Stratton M., Stubbington M. J. T., Theis F. J., Uhlen M., van Oudenaarden A., Wagner A., Watt F., Weissman J., Wold B., Xavier R., Yosef N., eLife, 2017, 6, e27041 |
| 3 | Ahmad R., Budnik B., Anal. Bioanal. Chem., 2023, 415(28), 6889—6899 |
| 4 | Liu Y., Beyer A., Aebersold R., Cell, 2016, 165(3), 535—550 |
| 5 | Labib M., Kelley S. O., Nat. Rev. Chem., 2020, 4(3), 143—158 |
| 6 | Zhu Y., Scheibinger M., Ellwanger D. C., Krey J. F., Choi D., Kelly R. T., Heller S., Barr⁃Gillespie P. G., eLife, 2019, 8, e50777 |
| 7 | Nelep C., Eberhardt J., Cytom. A, 2018, 93(12), 1267—1270 |
| 8 | Wang Y., Wang D. F., Wang H. F., Wang J. W., Pan J. Z., Guo X. G., Fang Q., Talanta, 2021, 226, 122136 |
| 9 | Wang Y., Guan Z. Y., Shi S. W., Jiang Y. R., Zhang J., Yang Y., Wu Q., Wu J., Chen J. B., Ying W. X., Xu Q. Q., Fan Q. X., Wang H. F., Zhou L., Wang L., Fang J., Pan J. Z., Fang Q., Nat. Commun., 2024, 15(1), 1279 |
| 10 | Nagamoto⁃Combs K., Animal Models of Allergic Disease: Methods and Protocols, Springer US, New York, 2021, 169—182 |
| 11 | Adan A., Alizada G., Kiraz Y., Baran Y., Nalbant A., Crit. Rev. Biotechnol., 2017, 37(2), 163—176 |
| 12 | Gross A., Schoendube J., Zimmermann S., Steeb M., Zengerle R., Koltay P., Int. J. Mol. Sci., 2015, 16(8), 16897—16919. |
| 13 | Vallone V. F., Telugu N. S., Fischer I., Miller D., Schommer S., Diecke S., Stachelscheid H., Curr. Protoc. Stem Cell Biology, 2020, 55(1), e123 |
| 14 | Song Y., Lin J. M., Sci. Sin. Chim., 2023, 53(8), 1472—1493 |
| 宋扬, 林金明. 中国科学: 化学, 2023, 53(8), 1472—1493 | |
| 15 | Pan T., Wu Y. Y., Guo G. S., Wang X. Y., Chin. J. Anal. Chem., 2023, 51(6), 934—944 |
| 潘婷, 武园园, 郭广生, 汪夏燕. 分析化学, 2023, 51(6), 934—944 | |
| 16 | Gebreyesus S. T., Siyal A. A., Kitata R. B., Chen E. S. W., Enkhbayar B., Angata T., Lin K. I., Chen Y. J., Tu H. L., Nat. Commun., 2022, 13(1), 37 |
| 17 | Hu S., Ye J., Shi S., Yang C., Jin K., Hu C., Wang D., Ma H., Anal. Chem., 2023, 95(17), 6905—6914 |
| 18 | Yang Z., Jin K., Chen Y., Liu Q., Chen H., Hu S., Wang Y., Pan Z., Feng F., Shi M., Xie H., Ma H., Zhou H., JACS Au, 2024, 4(5), 1811—1823 |
| 19 | Guo W., Hu Y., Qian J., Zhu L., Cheng J., Liao J., Fan X., J. Genet. Genom., 2023, 50(9), 641—651 |
| 20 | Espina V., Heiby M., Pierobon M., Liotta L. A., Expert. Rev. Mol. Diagn., 2007, 7(5), 647—657 |
| 21 | Mund A., Coscia F., Kriston A., Hollandi R., Kovács F., Brunner A. D., Migh E., Schweizer L., Santos A., Bzorek M., Naimy S., Rahbek⁃Gjerdrum L. M., Dyring⁃Andersen B., Bulkescher J., Lukas C., Eckert M. A., Lengyel E., Gnann C., Lundberg E., Horvath P., Mann M., Nat. Biotechnol., 2022, 40(8), 1231—1240 |
| 22 | Rosenberger F. A., Thielert M., Strauss M. T., Schweizer L., Ammar C., Mädler S. C., Metousis A., Skowronek P., Wahle M., Madden K., Gote⁃Schniering J., Semenova A., Schiller H. B., Rodriguez E., Nordmann T. M., Mund A., Mann M., Nat. Methods, 2023, 20(10), 1530—1536 |
| 23 | Liu J., Liu Z., Chin. J. Chromatogr., 2016, 34(1), 1154—1160 |
| 刘佳, 刘震. 色谱, 2016, 34(1), 1154—1160 | |
| 24 | Chen Q., Yan G., Gao M., Zhang X., Anal. Chem., 2015, 87(13), 6674—6680 |
| 25 | Shao X., Wang X., Guan S., Lin H., Yan G., Gao M., Deng C., Zhang X., Anal. Chem., 2018, 90(23), 14003—14010 |
| 26 | He Y., Yuan H., Liang Y., Liu X., Zhang X., Ji Y., Zhao B., Yang K., Zhang J., Zhang S., Zhang Y., Zhang L., Chem. Sci., 2023, 14(46), 13495—13502 |
| 27 | Johnson K. R., Gao Y., Greguš M., Ivanov A. R., Anal. Chem., 2022, 94(41), 14358—14367 |
| 28 | Marie A. L., Gao Y., Ivanov A. R., Nat. Commun., 2024, 15(1), 3847 |
| 29 | Zhu Y., Zhang Y. X., Cai L. F., Fang Q., Anal. Chem., 2013, 85(14), 6723—6731 |
| 30 | Li Z. Y., Huang M., Wang X. K., Zhu Y., Li J. S., Wong C. C. L., Fang Q., Anal. Chem., 2018, 90(8), 5430—5438 |
| 31 | Zhu Y., Piehowski P. D., Zhao R., Chen J., Shen Y., Moore R. J., Shukla A. K., Petyuk V. A., Campbell⁃Thompson M., Mathews C. E., Smith R. D., Qian W. J., Kelly R. T., Nat. Commun., 2018, 9(1), 882 |
| 32 | Liang Y., Acor H., McCown M. A., Nwosu A. J., Boekweg H., Axtell N. B., Truong T., Cong Y., Payne S. H., Kelly R. T., Anal. Chem., 2021, 93(3), 1658—1666 |
| 33 | Woo J., Williams S. M., Markillie L. M., Feng S., Tsai C. F., Aguilera⁃Vazquez V., Sontag R. L., Moore R. J., Hu D., Mehta H. S., Cantlon⁃Bruce J., Liu T., Adkins J. N., Smith R. D., Clair G. C., Pasa⁃Tolic L., Zhu Y., Nat. Commun., 2021, 12(1), 6246 |
| 34 | Specht H., Harmange G., Perlman D. H., Emmott E., Niziolek Z., Budnik B., Slavov N., bioRxiv, 2018, 399774[2018⁃08⁃25]. https: //doi.org/10.1101/399774 |
| 35 | Leduc A., Huffman R. G., Cantlon J., Khan S., Slavov N., Genome Biol., 2022, 23(1), 261 |
| 36 | Li Y., Li H., Xie Y., Chen S., Qin R., Dong H., Yu Y., Wang J., Qian X., Qin W., Anal. Chem., 2021, 93(42), 14059—14067 |
| 37 | Brunner A. D., Thielert M., Vasilopoulou C., Ammar C., Coscia F., Mund A., Hoerning O. B., Bache N., Apalategui A., Lubeck M., Richter S., Fischer D. S., Raether O., Park M. A., Meier F., Theis F. J., Mann M., Mol. Syst. Biology, 2022, 18(3), e10798 |
| 38 | Krieger J. R., Wybenga⁃Groot L. E., Tong J., Bache N., Tsao M. S., Moran M. F., J. Proteome Res., 2019, 18(5), 2346—2353 |
| 39 | Hartlmayr D., Ctortecka C., Seth A., Mendjan S., Tourniaire G., Mechtler K., bioRxiv, 2021, 2021.04.14.439828. https: //doi. org/10.1101/2021.04.14.439828 |
| 40 | Ye Z., Sabatier P., van der Hoeven L., Phlairaharn T., Hartlmayr D., Izaguirre F., Seth A., Joshi H. J., Bekker⁃Jensen D. B., Bache N., Olsen J. V., bioRxiv, 2023, 2023.11.27.568953[2023⁃11⁃28]. https: //doi.org/10.1101/2023.11.27.568953 |
| 41 | Ye Z., Sabatier P., Martin⁃Gonzalez J., Eguchi A., Lechner M., Østergaard O., Xie J., Guo Y., Schultz L., Truffer R., Bekker⁃Jensen D. B., Bache N., Olsen J. V., Nat. Commun., 2024, 15(1), 2474 |
| 42 | Wilson S. R., Olsen C., Lundanes E., Analyst, 2019, 144(24), 7090—7104 |
| 43 | Cong Y., Liang Y., Motamedchaboki K., Huguet R., Truong T., Zhao R., Shen Y., Lopez⁃Ferrer D., Zhu Y., Kelly R. T., Anal. Chem., 2020, 92(3), 2665—2671 |
| 44 | Cong Y., Motamedchaboki K., Misal S. A., Liang Y., Guise A. J., Truong T., Huguet R., Plowey E. D., Zhu Y., Lopez⁃Ferrer D., Kelly R. T., Chem. Sci., 2021, 12(3), 1001—1006 |
| 45 | Liang Y., Wang C., Liang Z., Zhang L., Zhang Y., Anal. Chem., 2022, 94(16), 6084—6088 |
| 46 | Xiang P., Zhu Y., Yang Y., Zhao Z., Williams S. M., Moore R. J., Kelly R. T., Smith R. D., Liu S., Anal. Chem., 2020, 92(7), 4711—4715 |
| 47 | Stejskal K., Op de Beeck J., Dürnberger G., Jacobs P., Mechtler K., Anal. Chem., 2021, 93(25), 8704—8710 |
| 48 | Petrosius V., Aragon⁃Fernandez P., Üresin N., Kovacs G., Phlairaharn T., Furtwängler B., Op De Beeck J., Skovbakke S. L., Goletz S., Thomsen S. F., Keller U. A. D., Natarajan K. N., Porse B. T., Schoof E. M., Nat. Commun., 2023, 14(1), 5910 |
| 49 | Matzinger M., Schmücker A., Yelagandula R., Stejskal K., Krššáková G., Berger F., Mechtler K., Mayer R. L., Nat. Commun., 2024, 15(1), 1019 |
| 50 | Qin S. J., Bai Y., Liu H. W., Chin. J. Chromatogr., 2021, 39(2), 142—151 |
| 秦少杰, 白玉, 刘虎威. 色谱, 2021, 39(2), 142—151 | |
| 51 | Tabb D. L., Vega⁃Montoto L., Rudnick P. A., Variyath A. M., Ham A. J. L., Bunk D. M., Kilpatrick L. E., Billheimer D. D., Blackman R. K., Cardasis H. L., Carr S. A., Clauser K. R., Jaffe J. D., Kowalski K. A., Neubert T. A., Regnier F. E., Schilling B., Tegeler T. J., Wang M., Wang P., Whiteaker J. R., Zimmerman L. J., Fisher S. J., Gibson B. W., Kinsinger C. R., Mesri M., Rodriguez H., Stein S. E., Tempst P., Paulovich A. G., Liebler D. C., Spiegelman C., J. Proteome Res., 2010, 9(2), 761—776 |
| 52 | Ludwig C., Gillet L., Rosenberger G., Amon S., Collins B. C., Aebersold R., Mol. Syst. Biology, 2018, 14(8), e8126 |
| 53 | Zhang F., Ge W., Ruan G., Cai X., Guo T., Proteomics, 2020, 20(17/18), 1900276 |
| 54 | Hou X. H., Zhou P. Y., Gong P. Y., Fu J. L., Liu C., Wang H. P., Prog. Biochem. Biophys., 2022, 49(12), 2364—2386 |
| 侯鑫行, 周丕宇, 宫鹏云, 付嘉乐, 刘超, 王海鹏. 生物化学与生物物理进展, 2022, 49(12), 2364—2386 | |
| 55 | Lou R., Shui W., Mol. Cell. Proteomics, 2024, 23(2), 100712 |
| 56 | Searle B. C., Swearingen K. E., Barnes C. A., Schmidt T., Gessulat S., Küster B., Wilhelm M., Nat. Commun., 2020, 11(1), 1548 |
| 57 | Phlairaharn T., Ye Z., Krismer E., Pedersen A. K., Pietzner M., Olsen J. V., Schoof E. M., Searle B. C., Anal. Chem., 2023, 95(26), 9881—9891 |
| 58 | Matzinger M., Müller E., Dürnberger G., Pichler P., Mechtler K., Anal. Chem., 2023, 95(9), 4435—4445 |
| 59 | Guzman U. H., Martinez⁃Val A., Ye Z., Damoc E., Arrey T. N., Pashkova A., Renuse S., Denisov E., Petzoldt J., Peterson A. C., Harking F., Østergaard O., Rydbirk R., Aznar S., Stewart H., Xuan Y., Hermanson D., Horning S., Hock C., Makarov A., Zabrouskov V., Olsen J. V., Nat. Biotechnol., 2024[2024⁃02⁃01]. https: //doi.org/10.1038/s41587⁃023⁃02099⁃7 |
| 60 | Bubis J. A., Arrey T. N., Damoc E., Delanghe B., Slovakova J., Sommer T. M., Kagawa H., Pichler P., Rivron N., Mechtler K., Matzinger M., bioRxiv, 2024, 2024.02.01.578358[2024⁃02⁃27]. https: //doi.org/10.1101/2024.02.01.578358 |
| 61 | Bekker⁃Jensen D. B., Martínez⁃Val A., Steigerwald S., Rüther P., Fort K. L., Arrey T. N., Harder A., Makarov A., Olsen J. V., Mol. Cell. Proteomics, 2020, 19(4), 716—729 |
| 62 | Reilly L., Lara E., Ramos D., Li Z., Pantazis C. B., Stadler J., Santiana M., Roberts J., Faghri F., Hao Y., Nalls M. A., Narayan P., Liu Y., Singleton A. B., Cookson M. R., Ward M. E., Qi Y. A., Cell Rep. Methods, 2023, 3(10), 100593 |
| 63 | Ridgeway M. E., Lubeck M., Jordens J., Mann M., Park M. A., Int. J. Mass Spectrom., 2018, 425, 22—35 |
| 64 | Vasilopoulou C. G., Sulek K., Brunner A. D., Meitei N. S., Schweiger⁃Hufnagel U., Meyer S. W., Barsch A., Mann M., Meier F., Nat. Commun., 2020, 11(1), 331 |
| 65 | Meier F., Brunner A. D., Frank M., Ha A., Bludau I., Voytik E., Kaspar⁃Schoenefeld S., Lubeck M., Raether O., Bache N., Aebersold R., Collins B. C., Röst H. L., Mann M., Nat. Methods, 2020, 17(12), 1229—1236 |
| 66 | Skowronek P., Thielert M., Voytik E., Tanzer M. C., Hansen F. M., Willems S., Karayel O., Brunner A. D., Meier F., Mann M., Mol. Cell. Proteomics, 2022, 21(9), 100279 |
| 67 | Szyrwiel L., Sinn L., Ralser M., Demichev V., bioRxiv, 2022, 2022.10.31.514544[2022⁃10⁃31]. https: //doi.org/10.1101/2022.10. 31.514544 |
| 68 | Moseley M. A., Hughes C. J., Juvvadi P. R., Soderblom E. J., Lennon S., Perkins S. R., Thompson J. W., Steinbach W. J., Geromanos S. J., Wildgoose J., Langridge J. I., Richardson K., Vissers J. P. C., J. Proteome Res., 2018, 17(2), 770—779 |
| 69 | Messner C. B., Demichev V., Bloomfield N., Yu J. S. L., White M., Kreidl M., Egger A. S., Freiwald A., Ivosev G., Wasim F., Zelezniak A., Jürgens L., Suttorp N., Sander L. E., Kurth F., Lilley K. S., Mülleder M., Tate S., Ralser M., Nat. Biotechnol., 2021, 39(7), 846—854 |
| 70 | Skowronek P., Krohs F., Lubeck M., Wallmann G., Itang E. C. M., Koval P., Wahle M., Thielert M., Meier F., Willems S., Raether O., Mann M., Mol. Cell. Proteomics, 2023, 22(2), 100489 |
| 71 | Distler U., Łącki M. K., Startek M. P., Teschner D., Brehmer S., Decker J., Schild T., Krieger J., Krohs F., Raether O., Hildebrandt A., Tenzer S., bioRxiv, 2023, 2023.01.30.526204[2023⁃02⁃02]. https: //doi.org/10.1101/2023.01.30.526204 |
| 72 | Tian X., Permentier H. P., Bischoff R., Mass Spectrom. Rev., 2023, 42(2), 546—576 |
| 73 | Li J., Van Vranken J. G., Pontano Vaites L., Schweppe D. K., Huttlin E. L., Etienne C., Nandhikonda P., Viner R., Robitaille A. M., Thompson A. H., Kuhn K., Pike I., Bomgarden R. D., Rogers J. C., Gygi S. P., Paulo J. A., Nat. Methods, 2020, 17(4), 399—404 |
| 74 | Ning X., Li Q., Zi J., Mei Z., Liu J., Zhang Y., Bi M., Ren Y., Liu X., Lv C., Yao H., Sun J., Rao F., Li S., Liu S., Anal. Chem., 2023, 95(13), 5788—5795 |
| 75 | Budnik B., Levy E., Harmange G., Slavov N., Genome Biol., 2018, 19(1), 161 |
| 76 | Petelski A. A., Emmott E., Leduc A., Huffman R. G., Specht H., Perlman D. H., Slavov N., Nat. Protoc., 2021, 16(12), 5398—5425 |
| 77 | Specht H., Emmott E., Petelski A. A., Huffman R. G., Perlman D. H., Serra M., Kharchenko P., Koller A., Slavov N., Genome Biol., 2021, 22(1), 50 |
| 78 | Derks J., Leduc A., Wallmann G., Huffman R. G., Willetts M., Khan S., Specht H., Ralser M., Demichev V., Slavov N., Nat. Biotechnol., 2023, 41(1), 50—59 |
| 79 | Kang U. B., Yeom J., Kim H., Lee C., J. Proteome Res., 2010, 9(7), 3750—3758 |
| 80 | Thielert M., Itang E. C. M., Ammar C., Rosenberger F. A., Bludau I., Schweizer L., Nordmann T. M., Skowronek P., Wahle M., Zeng W. F., Zhou X. X., Brunner A. D., Richter S., Levesque M. P., Theis F. J., Steger M., Mann M., Mol. Syst. Biology, 2023, 19(9), e11503 |
| 81 | Nesvizhskii A. I., J. Proteomics, 2010, 73(11), 2092—2123 |
| 82 | Mallick P., Kuster B., Nat. Biotechnol., 2010, 28(7), 695—709 |
| 83 | Noor Z., Ahn S. B., Baker M. S., Ranganathan S., Mohamedali A., Brief. Bioinform., 2021, 22(2), 1620—1638 |
| 84 | Tyanova S., Temu T., Cox J., Nat. Protoc., 2016, 11(12), 2301—2319 |
| 85 | Kong A. T., Leprevost F. V., Avtonomov D. M., Mellacheruvu D., Nesvizhskii A. I., Nat. Methods, 2017, 14(5), 513—520 |
| 86 | da Veiga Leprevost F., Haynes S. E., Avtonomov D. M., Chang H. Y., Shanmugam A. K., Mellacheruvu D., Kong A. T., Nesvizhskii A. I., Nat. Methods, 2020, 17(9), 869—870 |
| 87 | Chi H., Liu C., Yang H., Zeng W. F., Wu L., Zhou W. J., Wang R. M., Niu X. N., Ding Y. H., Zhang Y., Wang Z. W., Chen Z. L., Sun R. X., Liu T., Tan G. M., Dong M. Q., Xu P., Zhang P. H., He S. M., Nat. Biotechnol., 2018, 36(11), 1059—1061 |
| 88 | Boekweg H., Van Der Watt D., Truong T., Johnston S. M., Guise A. J., Plowey E. D., Kelly R. T., Payne S. H., J. Proteome Res., 2022, 21(1), 182—188 |
| 89 | Huffman R. G., Chen A., Specht H., Slavov N., J. Proteome Res., 2019, 18(6), 2493—2500 |
| 90 | Chen A. T., Franks A., Slavov N., Plos Comput. Biol., 2019, 15(7), e1007082 |
| 91 | Wang B., Wang Y., Chen Y., Gao M., Ren J., Guo Y., Situ C., Qi Y., Zhu H., Li Y., Guo X., Brief. Bioinform., 2022, 23(4), bbac214 |
| 92 | Yu F., Haynes S. E., Nesvizhskii A. I., Mol. Cell. Proteomics, 2021, 20, 100077 |
| 93 | Kalxdorf M., Müller T., Stegle O., Krijgsveld J., Nat. Commun., 2021, 12(1), 4787 |
| 94 | Tsou C. C., Avtonomov D., Larsen B., Tucholska M., Choi H., Gingras A. C., Nesvizhskii A. I., Nat. Methods, 2015, 12(3), 258—264 |
| 95 | Röst H. L., Rosenberger G., Navarro P., Gillet L., Miladinović S. M., Schubert O. T., Wolski W., Collins B. C., Malmström J., Malmström L., Aebersold R., Nat. Biotechnol., 2014, 32(3), 219—223 |
| 96 | Zhou X. X., Zeng W. F., Chi H., Luo C., Liu C., Zhan J., He S. M., Zhang Z., Anal. Chem., 2017, 89(23), 12690—12697 |
| 97 | Cox J., Nat. Biotechnol., 2023, 41(1), 33—43 |
| 98 | Gessulat S., Schmidt T., Zolg D. P., Samaras P., Schnatbaum K., Zerweck J., Knaute T., Rechenberger J., Delanghe B., Huhmer A., Reimer U., Ehrlich H. C., Aiche S., Kuster B., Wilhelm M., Nat. Methods, 2019, 16(6), 509—518 |
| 99 | Tiwary S., Levy R., Gutenbrunner P., Salinas Soto F., Palaniappan K. K., Deming L., Berndl M., Brant A., Cimermancic P., Cox J., Nat. Methods, 2019, 16(6), 519—525 |
| 100 | Yang Y., Liu X., Shen C., Lin Y., Yang P., Qiao L., Nat. Commun., 2020, 11(1), 146 |
| 101 | Lou R., Liu W., Li R., Li S., He X., Shui W., Nat. Commun., 2021, 12(1), 6685 |
| 102 | Lou R., Tang P., Ding K., Li S., Tian C., Li Y., Zhao S., Zhang Y., Shui W., iScience, 2020, 23(3), 100903 |
| 103 | Hao Y., Chen M., Huang X., Xu H., Wu P., Chen S., Anal. Chem., 2023, 95(37), 14077—14085 |
| 104 | Chen M., Zhu P., Wan Q., Ruan X., Wu P., Hao Y., Zhang Z., Sun J., Nie W., Chen S., Anal. Chem., 2023, 95(19), 7495—7502 |
| 105 | Zeng W. F., Zhou X. X., Willems S., Ammar C., Wahle M., Bludau I., Voytik E., Strauss M. T., Mann M., Nat. Commun., 2022, 13(1), 7238 |
| 106 | Yang Y., Lin L., Qiao L., Expert. Rev. Proteomics, 2021, 18(12), 1031—1043 |
| 107 | Rosenberger G., Bludau I., Schmitt U., Heusel M., Hunter C. L., Liu Y., MacCoss M. J., MacLean B. X., Nesvizhskii A. I., Pedrioli P. G. A., Reiter L., Röst H. L., Tate S., Ting Y. S., Collins B. C., Aebersold R., Nat. Methods, 2017, 14(9), 921—927 |
| 108 | Siyal A. A., Chen E. S. W., Chan H. J., Kitata R. B., Yang J. C., Tu H. L., Chen Y. J., Anal. Chem., 2021, 93(51), 17003—17011 |
| 109 | Searle B. C., Pino L. K., Egertson J. D., Ting Y. S., Lawrence R. T., MacLean B. X., Villén J., MacCoss M. J., Nat. Commun., 2018, 9(1), 5128 |
| 110 | Demichev V., Messner C. B., Vernardis S. I., Lilley K. S., Ralser M., Nat. Methods, 2020, 17(1), 41—44 |
| 111 | Demichev V., Szyrwiel L., Yu F., Teo G. C., Rosenberger G., Niewienda A., Ludwig D., Decker J., Kaspar⁃Schoenefeld S., Lilley K. S., Mülleder M., Nesvizhskii A. I., Ralser M., Nat. Commun., 2022, 13(1), 3944 |
| 112 | Yu F., Teo G. C., Kong A. T., Fröhlich K., Li G. X., Demichev V., Nesvizhskii A. I., Nat. Commun., 2023, 14(1), 4154 |
| 113 | Bruderer R., Bernhardt O. M., Gandhi T., Miladinović S. M., Cheng L. Y., Messner S., Ehrenberger T., Zanotelli V., Butscheid Y., Escher C., Vitek O., Rinner O., Reiter L., Mol. Cell. Proteomics, 2015, 14(5), 1400—1410 |
| 114 | Bekker⁃Jensen D. B., Bernhardt O. M., Hogrebe A., Martinez⁃Val A., Verbeke L., Gandhi T., Kelstrup C. D., Reiter L., Olsen J. V., Nat. Commun., 2020, 11(1), 787 |
| 115 | Muntel J., Gandhi T., Verbeke L., Bernhardt O. M., Treiber T., Bruderer R., Reiter L., Mol. Omics, 2019, 15(5), 348—360 |
| 116 | Tsai C. F., Zhao R., Williams S. M., Moore R. J., Schultz K., Chrisler W. B., Pasa⁃Tolic L., Rodland K. D., Smith R. D., Shi T., Zhu Y., Liu T., Mol. Cell. Proteomics, 2020, 19(5), 828—838 |
| 117 | Schoof E. M., Furtwängler B., Üresin N., Rapin N., Savickas S., Gentil C., Lechman E., Keller U. a. d., Dick J. E., Porse B. T., Nat. Commun., 2021, 12(1), 3341 |
| 118 | Orsburn B. C., Yuan Y., Bumpus N. N., Nat. Commun., 2022, 13(1), 7246 |
| 119 | Petrosius V., Aragon⁃Fernandez P., Arrey T. N., Üresin N., Furtwängler B., Stewart H., Denisov E., Petzoldt J., Peterson A. C., Hock C., Damoc E., Makarov A., Zabrouskov V., Porse B. T., Schoof E. M., bioRxiv, 2023, 2023.06.06.543943[2023⁃06⁃08]. https: //doi.org/10.1101/2023.06.06.543943 |
| 120 | Jiang Y. R., Zhu L., Cao L. R., Wu Q., Chen J. B., Wang Y., Wu J., Zhang T. Y., Wang Z. L., Guan Z. Y., Xu Q. Q., Fan Q. X., Shi S. W., Wang H. F., Pan J. Z., Fu X. D., Wang Y., Fang Q., Cell Rep., 2023, 42(11), 113455 |
| 121 | Wu J., Xu Q. Q., Jiang Y. R., Chen J. B., Ying W. X., Fan Q. X., Wang H. F., Wang Y., Shi S. W., Pan J. Z., Fang Q., Anal. Chem., 2024, 96(14), 5499—5508 |
| 122 | Gatto L., Aebersold R., Cox J., Demichev V., Derks J., Emmott E., Franks A. M., Ivanov A. R., Kelly R. T., Khoury L., Leduc A., MacCoss M. J., Nemes P., Perlman D. H., Petelski A. A., Rose C. M., Schoof E. M., Van Eyk J., Vanderaa C., Yates J. R., Slavov N., Nat. Methods, 2023, 20(3), 375—386 |
| 123 | Caufield J. H., Fu J., Wang D., Guevara⁃Gonzalez V., Wang W., Ping P., J. Proteome Res., 2021, 20(5), 2182—2186 |
| 124 | Wang F., Liu C., Li J., Yang F., Song J., Zang T., Yao J., Wang G., Nucleic Acids Res., 2024, 52(D1), D562⁃D571 |
| 125 | Li W., Yang F., Wang F., Rong Y., Liu L., Wu B., Zhang H., Yao J., Nat. Methods, 2024, 21(4), 623—634 |
| 126 | Wang F., Yang F., Huang L., Li W., Song J., Gasser R. B., Aebersold R., Wang G., Yao J., Nat. Mach. Intell., 2023, 5(11), 1236—1249 |
| [1] | 侯泽金, 李荣其, 李健, 冯怡宁, 靳茜茜, 孙俊红, 曹洁. 基于GC-MS和机器学习的深静脉血栓形成预测[J]. 高等学校化学学报, 2024, 45(9): 20240199. |
| [2] | 刘康, 潘荣容, 江德臣. 基于纳米电极的单细胞内生物分子电化学分析[J]. 高等学校化学学报, 2024, 45(5): 20240027. |
| [3] | 陈晓萍, 王旭潭, 刘宁, 汪庆祥, 倪建聪, 杨伟强, 林振宇. MOFs基微流控电化学芯片对多种重金属离子的实时在线检测[J]. 高等学校化学学报, 2024, 45(2): 20230395. |
| [4] | 董沛滢, 刘彤, 秦伟捷. RNA-蛋白质复合物规模化富集与鉴定新方法[J]. 高等学校化学学报, 2024, 45(11): 20240091. |
| [5] | 石倩, 刘冬梅, 方小泥, 刘宝红. 基于基质辅助激光解析质谱的高通量酪氨酸酶活性及抑制剂分析[J]. 高等学校化学学报, 2024, 45(11): 20240330. |
| [6] | 蒋龑, 陈妍琳, 宋高瑜, 陈炎炎, 白晶, 朱莹娣, 李娟. 细菌的蛋白质组成分析[J]. 高等学校化学学报, 2024, 45(11): 20240345. |
| [7] | 闫勇杰, 高文博, 鲁晨辉, 杨成, 徐姝婷. 基于微萃取-纳喷雾质谱技术的纳升脑脊液中咖啡多酚的检测[J]. 高等学校化学学报, 2024, 45(11): 20240327. |
| [8] | 张磊, 申华莉. 标志物研究中常用蛋白组学质谱方法的定量准确性评估[J]. 高等学校化学学报, 2024, 45(11): 20240311. |
| [9] | 曹婷, 舒伟康, 万晶晶. 等离子体复合材料辅助小分子代谢物的质谱半定量分析及鉴定[J]. 高等学校化学学报, 2024, 45(11): 20240325. |
| [10] | 张怡涵, 滑天宇, 侯士姣, 张洋洋, 殷丹, 姬向波, 张岩皓, 裴聪聪, 张书胜. 尖端Fe2O3纳米棒驱动的高性能质谱分析用于构建PM2.5暴露鼠的代谢指纹图谱[J]. 高等学校化学学报, 2024, 45(11): 20240376. |
| [11] | 靳莹, 张俊杰, 张毅欣, 袁悦, 韩珍珍. 外泌体分离和蛋白质组学分析的研究进展[J]. 高等学校化学学报, 2024, 45(11): 20240305. |
| [12] | 胡宇虹, 俞相明, 宋丽丽, 邢清和, 周峰. DEEP SEQ方法检测新生儿毛干蛋白质组动态变化[J]. 高等学校化学学报, 2024, 45(11): 20240326. |
| [13] | 许霞, 秦伟达, 李若萌, 王倩倩, 刘宁, 李功玉. 深度覆盖蛋白质组学质谱分析: 细胞蛋白提取方法的评估[J]. 高等学校化学学报, 2024, 45(11): 20240344. |
| [14] | 续红妹, 王梁臣, 闵乾昊. 面向小分子检测的MALDI MS基质研究进展[J]. 高等学校化学学报, 2024, 45(11): 20240285. |
| [15] | 黄玉滢, 于成鲲, 刘斯奇, 任艳. 利用无标记单细胞蛋白质组学方法构建小鼠外周血单个核细胞的细胞图谱[J]. 高等学校化学学报, 2024, 45(11): 20240355. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||