

高等学校化学学报 ›› 2019, Vol. 40 ›› Issue (12): 2512.doi: 10.7503/cjcu20190444
收稿日期:2019-08-07
出版日期:2019-12-04
发布日期:2019-12-04
通讯作者:
孙仕勇
E-mail:shysun@swust.edu.cn
基金资助:
Xiaoyu FAN,Ke WANG,Shiyong SUN( ),Biaobiao MA,Rui LÜ
),Biaobiao MA,Rui LÜ
Received:2019-08-07
Online:2019-12-04
Published:2019-12-04
Contact:
Shiyong SUN
E-mail:shysun@swust.edu.cn
Supported by:摘要:
以铁氨基黏土(Fe-aminoclay)为载体, 1-(3-二甲氨基丙基)-3-乙基碳二亚胺盐酸盐(EDC)为共价交联剂, 构筑了铁氨基黏土纳米结构脂肪酶催化剂(Feclay-lipase). 利用X射线衍射(XRD)、 透射电子显微镜(TEM)和傅里叶变换红外光谱(FTIR)等技术对Feclay-lipase进行了表征, 并通过酶动力学对比研究了游离脂肪酶和Feclay-lipase的酶学特性. 结果表明, Fe-aminoclay的载酶量为414.4 mg/g, 固定化效率可达82.88%, Feclay-lipase的酶活较游离酶提高了3倍, 最适反应温度提高了10 ℃, 最适反应 pH向碱性偏移, 储存稳定性更好, 在4 ℃下贮存30 d后其酶活无明显减弱.
中图分类号:
TrendMD:
范小雨,王可,孙仕勇,马彪彪,吕瑞. 铁氨基黏土纳米结构脂肪酶的构筑及催化特性. 高等学校化学学报, 2019, 40(12): 2512.
Xiaoyu FAN,Ke WANG,Shiyong SUN,Biaobiao MA,Rui LÜ. Construction and Catalytic Performances of Fe-aminoclay Nanostructured Lipase †. Chem. J. Chinese Universities, 2019, 40(12): 2512.
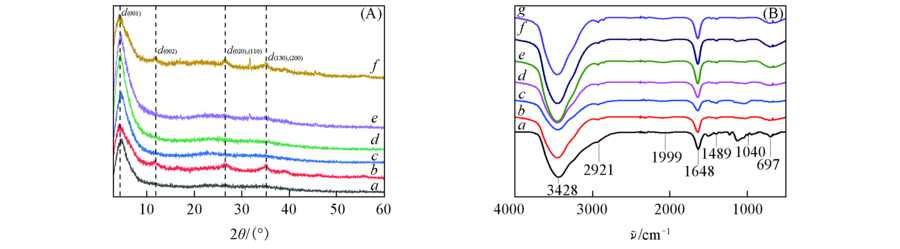
Fig.1 XRD patterns(A) and FTIR spectra(B) of Fe-aminoclay and Feclay-lipase with various lipase loadings (A) a. Fe-aminoclay; b. Feclay-10lipase; c. Feclay-50lipase; d. Feclay-100lipase; e. Feclay-150lipase; f. Feclay-200lipase. (B) a. Fe-aminoclay; b. lipase; c. Feclay-10lipase; d. Feclay-50lipase; e. Feclay-100lipase; f. Feclay-150lipase; g. Feclay-200lipase.
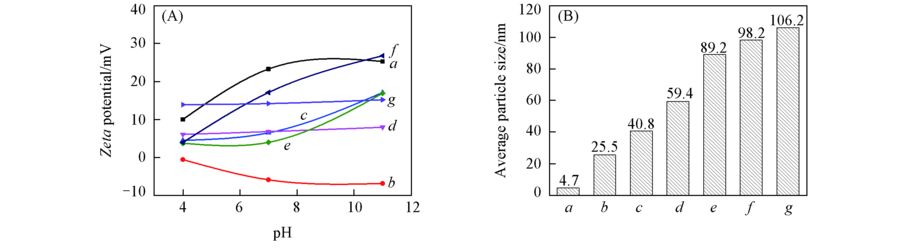
Fig.3 Zeta potentials(A) and average particle sizes(B) of Fe-aminoclay, lipase and Feclay-lipase a. Fe-aminoclay; b. lipase; c. Feclay-10lipase; d. Feclay-50lipase; e. Feclay-100lipase; f. Feclay-150lipase; g. Feclay-200lipase.

Fig.5 TEM images of Fe-aminoclay(A) and Feclay-lipase(B), SEM images of Fe-aminoclay(C), Feclay-150lipase(D) and the selected area in (D) image(E) and contact angle images of Fe-aminoclay(F), lipase(G) and Feclay-150lipase(H)
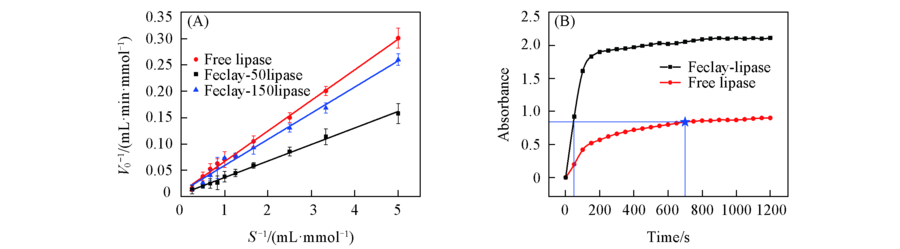
Fig.9 Lineweaver-Burk plots of the free lipase and Feclay-lipase(A), absorbance at the peak position for p-NP(410 nm) as a function of time for two different catalytic systems(B)
| Sample | R2 | Km/mmol | Vmax/(mmol·mL-1·min -1) |
|---|---|---|---|
| Free lipase | 0.997 | 5.35 | 99 |
| Feclay-50lipase | 0.996 | 6.98 | 217.39 |
| Feclay-150lipase | 0.996 | 7.84 | 175.42 |
Table 1 Kinetic parameters of the free and the Feclay-lipase
| Sample | R2 | Km/mmol | Vmax/(mmol·mL-1·min -1) |
|---|---|---|---|
| Free lipase | 0.997 | 5.35 | 99 |
| Feclay-50lipase | 0.996 | 6.98 | 217.39 |
| Feclay-150lipase | 0.996 | 7.84 | 175.42 |
| [1] |
Madhava A., Sindhu R., Binod P., Sukumaran R. K., Pandey A ., Bioresource. Technol., 2017,245, 1304— 1313
doi: 10.1016/j.biortech.2017.05.031 URL |
| [2] |
Kirk O., Borchert T. V., Fuglsang C. C ., Curr. Opin. Biotech., 2002,13, 345— 351
doi: 10.1016/s0958-1669(02)00328-2 URL pmid: 12323357 |
| [3] | Patel N., Rai D., Shivam S., Shahane S., Mishra U., Recent Pat . Biotechnol., 2018,13, 45— 56 |
| [4] |
Burton S. G., Cowan D. A., Woodley J. M ., Nat. Biotechnol., 2002,20, 37— 45
doi: 10.1038/nbt0102-37 URL pmid: 11753360 |
| [5] |
Adlercreutz P ., Chem. Soc. Rev., 2013,42, 6406— 6436
doi: 10.1039/c3cs35446f URL pmid: 23403895 |
| [6] |
Shi J., Wang X., Zhang W., Jiang Z., Liang Y., Zhu Y., Zhang C ., Adv. Funct. Mater., 2013,23, 1450— 1458
doi: 10.1002/adfm.201202068 URL |
| [7] |
Datta K. K. R., Achari A., Eswaramoorthy M ., J. Mater. Chem. A, 2013,1, 6707— 6718
doi: 10.1039/c3ta00100h URL |
| [8] |
Bilal M., Iqbal H. M. N ., Coordin. Chem. Rev., 2019,388, 1— 23
doi: 10.1016/j.ccr.2019.02.024 URL |
| [9] |
Ren S., Li C., Jiao X., Jia S., Jiang Y., Bilal M., Cui J ., Chem. Eng. J., 2019,373, 1254— 1278
doi: 10.1016/j.cej.2019.05.141 URL |
| [10] |
Rodrigues R. C., Virgen Ortiz J. J., Dos Santos J. C. S., Berenguer Murcia A., Alcantara A. R., Barbosa O., Ortiz C., Fernandez Lafuente R ., Biotechnol. Adv., 2019,37, 746— 770
doi: 10.1016/j.biotechadv.2019.04.003 URL pmid: 30974154 |
| [11] |
Ramos E. Z., Júnior R. H. M., de Castro P. F., Tardioli P. W., Mendes A. A., Fernandéz Lafuente R., Hirata D. B ., J. Mol. Catal. B:Enzym., 2015,118, 43— 51
doi: 10.1016/j.molcatb.2015.05.009 URL |
| [12] |
Lin S., Sun S., Wang K., Shen K., Ma B., Ren Y., Fan X ., Nanomaterials, 2018,8, 127— 135
doi: 10.3390/nano8020127 URL |
| [13] |
Sun S., Li M., Dong F., Wang S., Tian L., Mann S ., Small, 2016,12, 1920— 1927
doi: 10.1002/smll.201600243 URL pmid: 26923794 |
| [14] |
Jiang Y., Liu X., Chen Y., Zhou L., He Y., Ma L., Gao J ., Bioresource. Technol., 2014,153, 278— 283
doi: 10.1016/j.biortech.2013.12.001 URL |
| [15] |
Zhang C., Dong X., Guo Z., Sun Y ., J. Colloid. Interf. Sci., 2018,519, 145— 153
doi: 10.1016/j.jcis.2018.02.039 URL pmid: 29494877 |
| [16] |
Jamwal S., Kumar D., Ranote S., Chauhan G. S ., J. Nanosci. Nanotechnol., 2019,19, 7205— 7214
doi: 10.1166/jnn.2019.16667 URL pmid: 31039877 |
| [17] |
Xie W. L., Zang X. Z ., Food Chem., 2017,227, 397— 403
doi: 10.1016/j.foodchem.2017.01.082 URL pmid: 28274449 |
| [18] |
Zhu J., Sun G ., React. Funct. Polym., 2012,72, 839— 845
doi: 10.1016/j.reactfunctpolym.2012.08.001 URL |
| [19] |
Zdarta J., Meyer A. S., Jesionowski T., Pinelo M ., Catalysts, 2018,8, 92— 118
doi: 10.1186/1471-2148-8-92 URL pmid: 18366743 |
| [20] | Mao C ., Small, 2005,1, 356— 356 |
| [21] |
Kim J., Grate J. W., Wang P ., Chem. Eng. Sci., 2006,61, 1017— 1026
doi: 10.1016/j.ces.2005.05.067 URL |
| [22] |
Hong T., Liu W., Li M., Chen C ., Anal. Chim. Acta, 2019,1067, 31— 47
doi: 10.1016/j.aca.2019.02.031 URL pmid: 31047147 |
| [23] |
Liu X., Fang Y., Xu Y., Yong L., Wang C ., Chem. Eng. J., 2018,336, 456— 464
doi: 10.1016/j.cej.2017.12.048 URL |
| [24] |
Rege K., Raravikar N. R., Kim D. Y., Schadler L. S., Ajayan P. M., Dordick J. S ., Nano Lett., 2015,3, 829— 832
doi: 10.1021/nl034131k URL |
| [25] |
Wang Z. G., Wan L. S., Liu Z. M., Huang X. J., Xu Z. K ., J. Mol. Catal. B: Enzym., 2009,56, 189— 195
doi: 10.1016/j.molcatb.2008.05.005 URL |
| [26] |
Zhang C., Liu Y., Sun Y ., Biochem. Eng. J., 2019,146, 124— 131
doi: 10.1016/j.bej.2019.03.012 URL |
| [27] | Xiang X., Wan X., Suo H., Hu Y ., Acta Phys-Chim. Sin., 2018,34, 99— 107 |
| [28] |
Tzialla A. A., Pavlidis I. V., Felicissimo M. P., Rudolf P., Gournis D., Stamatis H ., Bioresource Technol., 2010,101, 1587— 1594
doi: 10.1016/j.biortech.2009.10.023 URL pmid: 19910187 |
| [29] | Öztürk H., Pollet E., Phalip V., Güvenilir Y., Avérous L ., Polymers-Basel., 2016,8, 416— 432 |
| [30] |
Chen N., Zhang C., Liu Y., Dong X., Sun Y ., Biochem. Eng. J., 2019,145, 137— 144
doi: 10.1016/j.bej.2019.02.018 URL |
| [31] |
Miao C., Yang L., Wang Z., Luo W., Li H., Lv P., Yuan Z ., Fuel, 2018,224, 774— 782
doi: 10.1016/j.fuel.2018.02.149 URL |
| [32] |
Bui V. K. H., Park D., Lee Y. C ., Chem. Eng. J., 2018,336, 757— 772
doi: 10.1016/j.cej.2017.12.052 URL |
| [33] |
Patil A. J., Muthusamy E., Mann S ., J. Mater. Chem., 2005,15, 3838— 3843
doi: 10.1039/b504288g URL |
| [34] |
Lee Y. C., Kim M. I., Woo M. A., Park H. G., Han J. I ., Biosens. Bioelectron., 2013,42, 373— 378
doi: 10.1016/j.bios.2012.10.092 URL pmid: 23211453 |
| [35] |
Bradford M. M ., Anal. Biochem., 1976,72, 248— 254
doi: 10.1006/abio.1976.9999 URL pmid: 942051 |
| [36] |
Shah E., Mahapatra P., Bedekar A. V., Soni H. P ., RSC Adv., 2015,5, 26291— 26300
doi: 10.1039/C5RA02249E URL |
| [37] |
Jing G., Yun W., Du Y., Zhou L., Ying H., Li M., Yin L., Kong W., Jiang Y ., Chem. Eng. J., 2017,317, 175— 186
doi: 10.1016/j.cej.2017.02.012 URL |
| [38] |
Patil A. J., Mann S ., J. Mater. Chem., 2008,18, 4605— 4615
doi: 10.1039/b805653f URL |
| [39] |
Lebeau B., Brendlé J., Marichal C., Patil A. J., Muthusamy E., Mann S ., J. Nanosci. Nanotechnol., 2006,6, 352— 359
doi: 10.1166/jnn.2006.910 URL pmid: 16573032 |
| [40] |
Lee Y. C., Kim E. J., Dong A. K., Yang J. W ., J. Hazard. Mater., 2011,196, 101— 108
doi: 10.1016/j.jhazmat.2011.08.077 URL |
| [41] |
Han H. K., Lee Y. C., Lee M. Y., Patil A. J., Shin H. J ., ACS Appl. Mater. Interfaces, 2011,3, 2564— 2572
doi: 10.1021/am200406k URL pmid: 21609130 |
| [42] |
Dong H., Li J., Li Y., Hu L., Luo D ., Chem. Eng. J., 2012,181/182, 590— 596
doi: 10.1016/j.cej.2011.11.095 URL |
| [43] |
Hwang Y., Lee Y. C., Mines P. D., Yun S. H., Andersen H. R ., Appl. Catal. B, 2014,147, 748— 755
doi: 10.1016/j.apcatb.2013.10.017 URL |
| [44] |
Mateo C., Palomo J. M., Fernandez Lorente G., Guisan J. M., Fernandez Lafuente R ., Enzyme. Microb. Tech., 2007,40, 1451— 1463
doi: 10.1016/j.enzmictec.2007.01.018 URL |
| [45] |
Barriuso J., Vaquero M. E., Prieto A., Martínez M.J ., Biotechnol. Adv., 2016,34, 874— 885
doi: 10.1016/j.biotechadv.2016.05.004 URL pmid: 27188926 |
| [46] |
Liu J., Guo H., Zhou Q., Wang J., Lin B., Zhang H., Gao Z., Xia C., Zhou X ., J. Mol. Catal. B: Enzym., 2013,96, 96— 102
doi: 10.1016/j.molcatb.2013.06.013 URL |
| [47] |
Miranda M., Urioste D., Andrade Souza L. T., Mendes A. A., de Castro H. F ., Enzyme Res., 2011,2011, 216435
doi: 10.4061/2011/216435 URL pmid: 21876790 |
| [48] |
Zou B., Hu Y., Cui F., Jiang L., Yu D., Huang H ., J. Colloid Interf. Sci., 2014,417, 210— 216
doi: 10.1016/j.jcis.2013.11.029 URL pmid: 24407679 |
| [49] |
Wang J., Zhao G., Jing L., Peng X., Li Y ., Biochem. Eng. J., 2015,98, 75— 83
doi: 10.1016/j.bej.2014.11.013 URL |
| [50] |
Yong Y., Bai Y. X., Li Y. F., Lin L., Cui Y. J., Xia C. G ., Process Biochem., 2008,43, 1179— 1185
doi: 10.1016/j.procbio.2008.05.019 URL |
| [51] |
Reshmi R., Sugunan S ., J. Mol. Catal. B: Enzym., 2013,97, 36— 44
doi: 10.1016/j.molcatb.2013.04.003 URL |
| [52] | Mendes A. A., Barbosa B. C. M., Silva M. L. C. P. D., Castro H. F. D ., Biocatalysis, 2009,25, 393— 400 |
| [1] | 李柳, 孙仕勇, 吕瑞, GOLUBEVYevgeny Aleksandrovich, 王可, 董发勤, 段涛, KOTOVAOlga Borisovna, KOTOVAElena Leonidovna. 铁氨基黏土-葡萄糖氧化酶纳米复合催化剂的构筑及多酶级联反应研究[J]. 高等学校化学学报, 2021, 42(3): 803. |
| [2] | 王欢, 所金泉, 王春艳, 王润伟. 氨基化树枝状介孔二氧化硅固定葡萄糖氧化酶用于检测葡萄糖[J]. 高等学校化学学报, 2020, 41(8): 1731. |
| [3] | 高霞,潘会宾,乔成芳,陈凤英,周元,杨文华. 基于多级孔金属有机骨架构筑HRP固定化酶反应器及其染料降解应用[J]. 高等学校化学学报, 2020, 41(7): 1591. |
| [4] | 吴蓉, 董其惠, 孙伊伊, 苏二正. 吸附-纤维素覆膜联合固定化酶[J]. 高等学校化学学报, 2019, 40(9): 1888. |
| [5] | 宋艺超, 胡满成, 李淑妮, 翟全国, 蒋育澄. 基于中心辐射树枝状介孔二氧化硅构筑CPO固定化酶反应器及应用[J]. 高等学校化学学报, 2019, 40(9): 1805. |
| [6] | 张璋, 王栋, 王晓雷, 徐岩. 华根霉脂肪酶有机相酯合成活性的重塑[J]. 高等学校化学学报, 2019, 40(4): 747. |
| [7] | 杨一涵, 王栋, 张璋, 徐岩. 表面活性剂对华根霉脂肪酶有机相酯合成活性的激活[J]. 高等学校化学学报, 2018, 39(9): 1948. |
| [8] | 张敏, 张祎, 李成涛, 覃家祥, 高让, 马晓宁, 邱建辉. 亲水改性PBS基共聚物的N435酶降解差异性及分子模拟[J]. 高等学校化学学报, 2015, 36(3): 568. |
| [9] | 张敏, 荆晶晶, 李成涛, 王慧, 马晓燕. 结合分子模拟探讨不同醚键PBS基共聚物的性能与酶催化降解[J]. 高等学校化学学报, 2014, 35(12): 2706. |
| [10] | 陈晨, 史倩, 陈军辉, 张茹潭, 李鑫, 郑立, 王小如. 基于固定化酶的乙酰胆碱酯酶抑制剂体外筛选模型的建立[J]. 高等学校化学学报, 2013, 34(5): 1121. |
| [11] | 胡燚, 杨姣, 唐苏苏, 初旭明, 邹彬, 黄和. Burkholderia Cepacia脂肪酶在氨基功能化离子液体修饰的SBA-15上的共价固定[J]. 高等学校化学学报, 2013, 34(5): 1195. |
| [12] | 陈锥, 陶益, 王怡, 王毅. 磁珠富集与液相色谱-质谱联用结合筛选枳壳中脂肪酶抑制剂[J]. 高等学校化学学报, 2012, 33(12): 2692. |
| [13] | 王周俊 李翔 王琛琛 唐颐 张亚红. 纳米沸石修饰微通道反应器内固定化酶催化的水解反应动力学[J]. 高等学校化学学报, 2011, 32(3): 753. |
| [14] | 张敏, 丁明亮, 张婷, 杨金明. 溶剂对脂肪酶降解高分子量PBS及改性共聚物的影响[J]. 高等学校化学学报, 2010, 31(3): 612. |
| [15] | 吴侠, 韩玉顺, 曹敏花, 胡长文, 任玲, 葛广路. α-胰凝乳蛋白酶在相转移Fe3O4纳米粒子表面的固定化[J]. 高等学校化学学报, 2010, 31(1): 11. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||